米国CDC、 FDA、 USDA- FSIS、 NCBIは次世代シークエンサー(NGS)を用いた食中毒細菌の全ゲノム解析のための提携を結び、Genome Trackrプロジェクトを2013年に開始しました。このプロジェクトが始まった2013年9月以降、原因食品を解明することができた食中毒事件数が急激に上昇しました。今回紹介する論文は、 Genome Trackrプロジェクト の開始前と開始後でリステリア菌による食中毒の原因究明がどれだけ改善されたかを紹介した論文です。
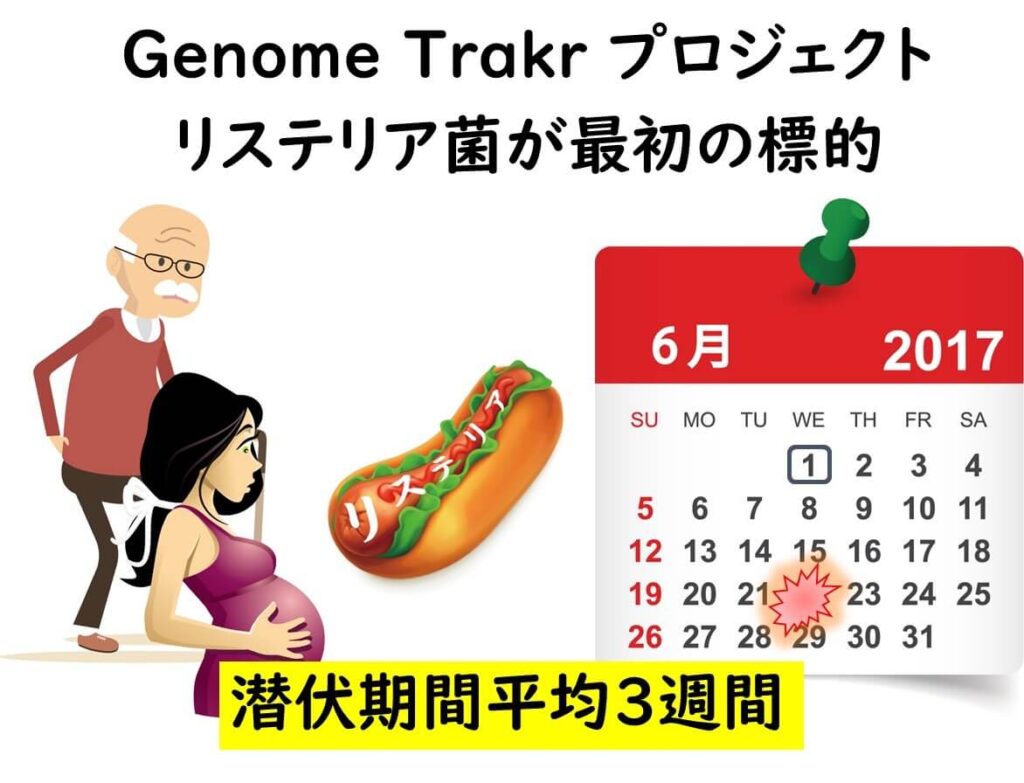
米国で細菌性食中毒の原因菌として心配されている代表格は、サルモネラ菌、リステリア菌、腸管出血性大腸菌などです。この中でGenomeTrakrプロジェクトの第1の標的候補としてはリステリア菌が選択されました。なぜでしょうか?
その理由は、リステリア菌の食中毒の原因究明が細菌性食中毒の中で最も難しいからです。なぜリステリア菌の食中毒の原因究明が難しいかというと、例えば妊婦がリステリア菌に汚染された食品を食べて流産などの重篤な症状を起こすまでの期間が約3週間もあるからです。たった一人の患者の食中毒すなわち散発事例の場合、患者にインタビューを行っても3週間前に 食べた食品を覚えていない場合がほとんどです。奇跡的にその食品を覚えていても、その食品が実際に患者の自宅の冷蔵庫に残っており、 そこから患者と同じタイプのリステリア菌が分離されない限り、食中毒の原因究明ができません。
※リステリア菌の特徴の基礎事項を確認したい方は下記の記事をご覧ください。
食中毒菌10種類の覚え方 ⑧リステリア菌
このようなリステリア菌の特殊な事情により、リステリア症感染は米国においても未解決事件として終わる場合が多かったわけです。この問題を何とか関係するために次世代シークエンサーによるリステリア菌の全ゲノム配列をデータベース化しようというゲノムトラッカープロジェクトが立ち上がったわけです。
以下に次世代シークエンサーを用いたゲノムトラッカープロジェクトがいかにこれまで未解決であったリステリア食中毒を解決に導いたかということを、2016年の初期段階での整理をした 米国 FDA や CDC の研究者たちが共同で報告した論文の内容を要点のみ紹介します。次世代シークエンサーによる微生物の全ゲノム解析とそのデータベース化がどのように食中毒菌の原因食品解析に力を発揮しているかということが示されています。
Genome Trakrプロジェクトの成功事例パターン①
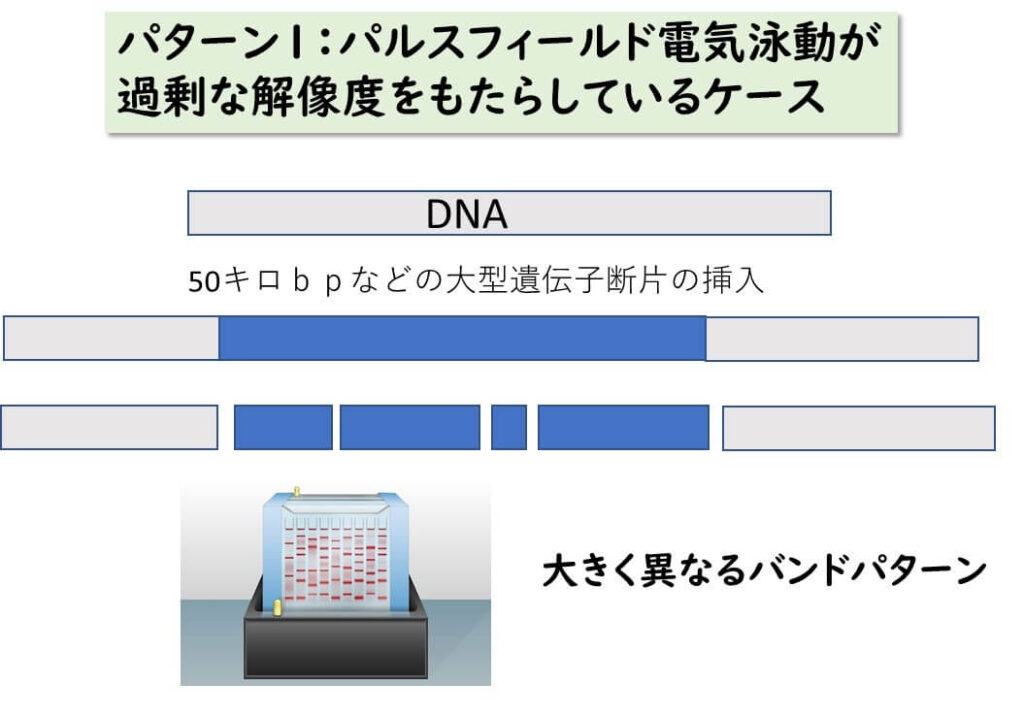
パルスフィールド電気泳動よりもwhole genome解析の方が解像度は高いです。しかし、その逆の場合も存在存在しているようです。whole genomeで解析すればほぼ同一のクローンに近いものであると判断できるにもかかわらず、パルスフィールド電気泳動は過度の解像度を提供してしまっているケースです。このような現象は、大きな遺伝子断片が水平伝播で挿入された場合などにおきます。
カンザス州の同一病院に入院している患者において3例のリステリア症患者が立て続けに起きました。ただしパルスフィールド電気泳動のバンドパターンでは3事例とも異なるバンドを示していました。このために病院の医師は、別の原因によるものだろうと考え、特段のアクションを起こしませんでした。次に4事例目が発生しました。4事例においては、2事例目と同一のパルスフィールド電気泳動パターンを示しました。そこで病院の医師はCDCに連絡をし、whole genome MLS T解析の依頼を行いました。

CDCがこれらの事例から分離されたリステリア菌株のwhole genome MLS T解析行いました。その結果、4株とも、whole genome MLS Tからは極めて近似の株であるという結論が得られました(4株のalleles違いは<18 alleles)
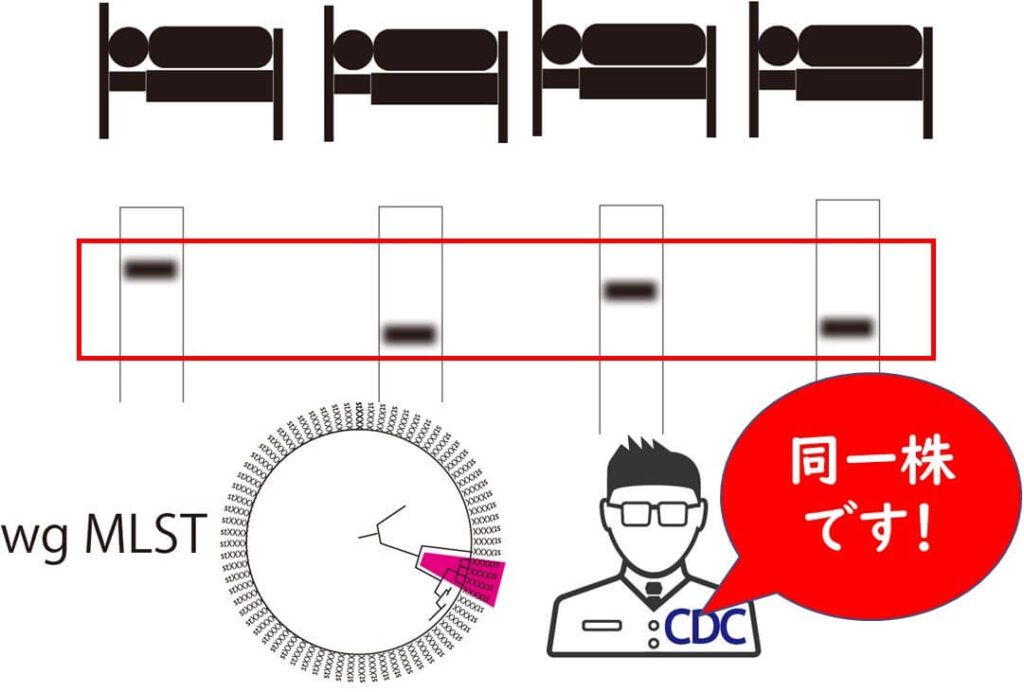
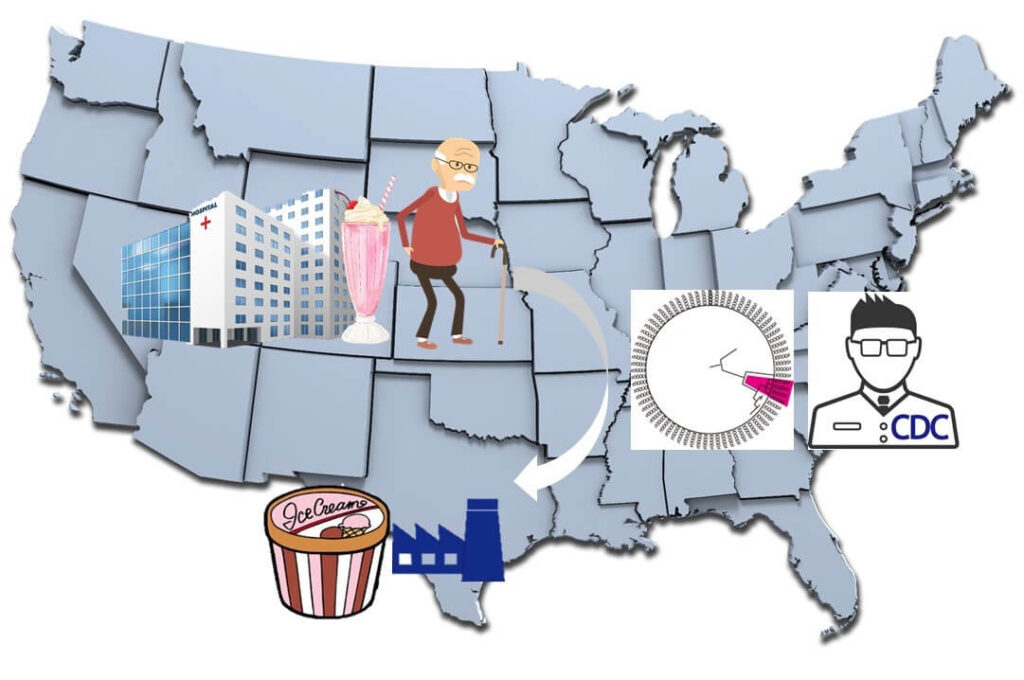
その後の疫学的な調査(患者はどこで何を食べたかなどの聞き取り調査)の結果、4人とも、病院の食堂において、アイスクリームから作られたミルクシェイクを飲んでいることが分かりました。

病院のミルクシェイクで使われていたアイスクリームの生産会社はテキサス州にありました。アイスクリーム工場を製品から分離したリステリア株と患者から分離されたリステリア株のwhole genomeMLS T解析を行った結果、両者は極めて近似していました(両者のalleles違いは<28 alleles)。以上から、これらのリステリア中毒はテキサス州にあるアイスクリーム工場が原因である結論されました。
Genome Trakrプロジェクトの成功事例パターン②
Geome Trakrプロジェクトはこれまで未解決であった食中毒事件を遡及的に解決する力を持っている。

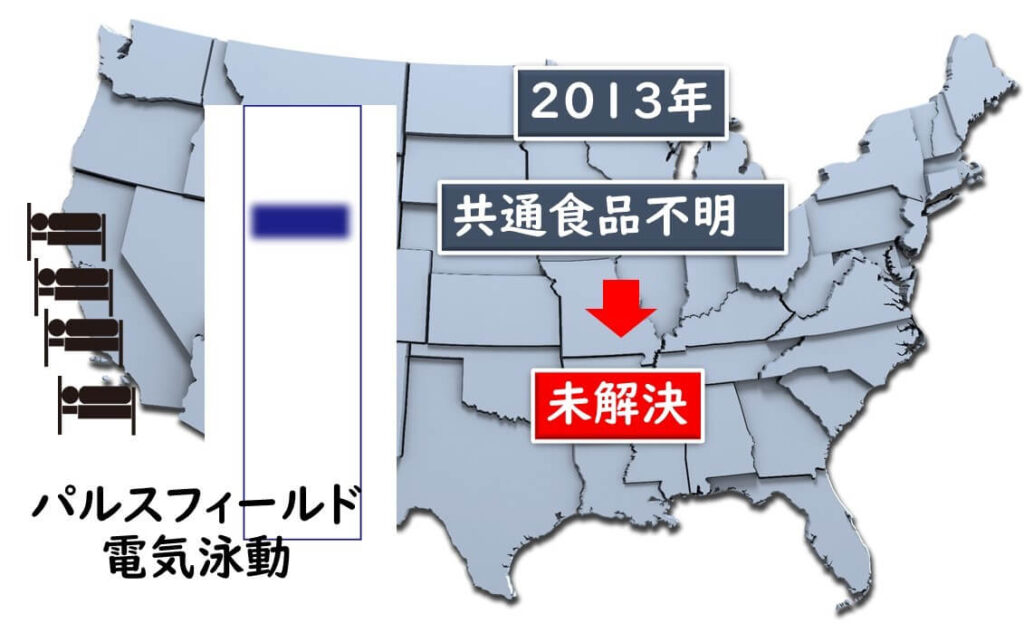
2013年、同一州において4人のリステリア患者が報告されました。パルスフィールド電気泳動パターンは同一パターンを示しました。したがって同一食品である原因であると推測されました。しかし現食品を見つけることができませんでした。結局この事例は、未解決食中毒事例として処理されまました。
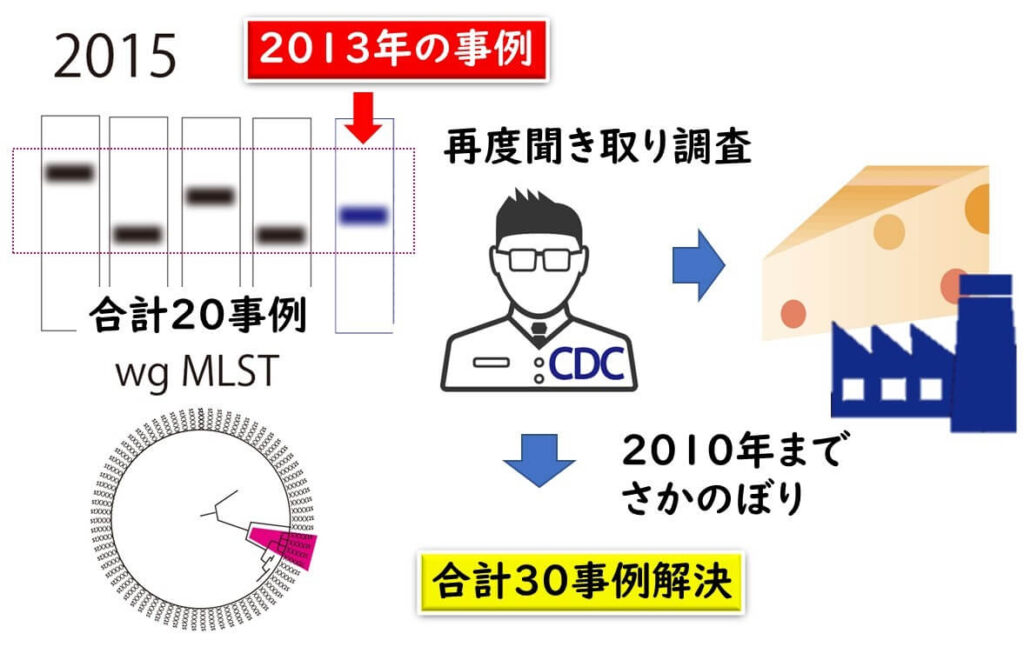
ところが、2015年8月、CDCは、2015年に発生していたリステリア症の臨床株を解析している過程で、whole genome MLS T解析行った結果、2年前の2013年の事例の5株も含めて合計20株はほぼ近似した株であることが判明しました(分析した菌株同士のallelesの違いは<25 alleles)。そこで、CDDの担当官は、さらに2010年まで未解決事件をさかのぼり解析してみました。
その結果、2010年まで遡った合計30事例について、同一工場で生産されたチーズが原因でリステリア食中毒がおきていたことを突き止めました。
Genome Trakrプロジェクトの成功事例パターン③
パルスフィールド電気泳動の解像度が低いために、実際は異なるoutbreakであるにもかかわらず同一のアウトブレークを疑ってしまうケースがあります。Geome Trakrプロジェクトでは、whole genome MLS T解析によって、このようなパルスフィールド電気泳動の低い解像度によって生じる問題を解決することができることを示しました。
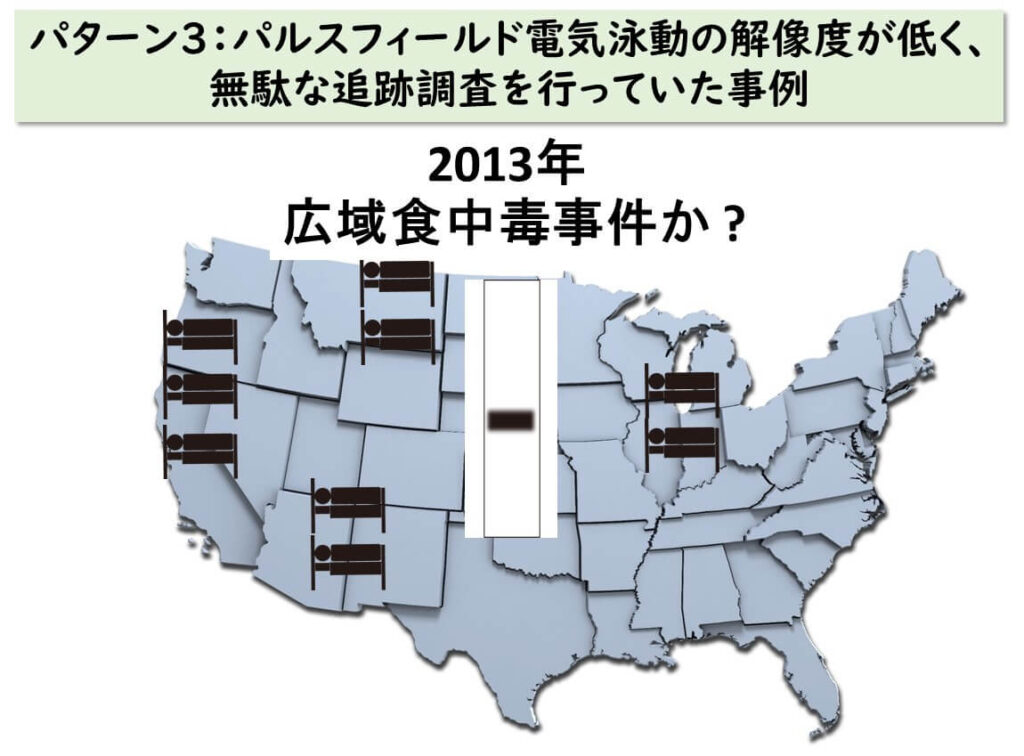
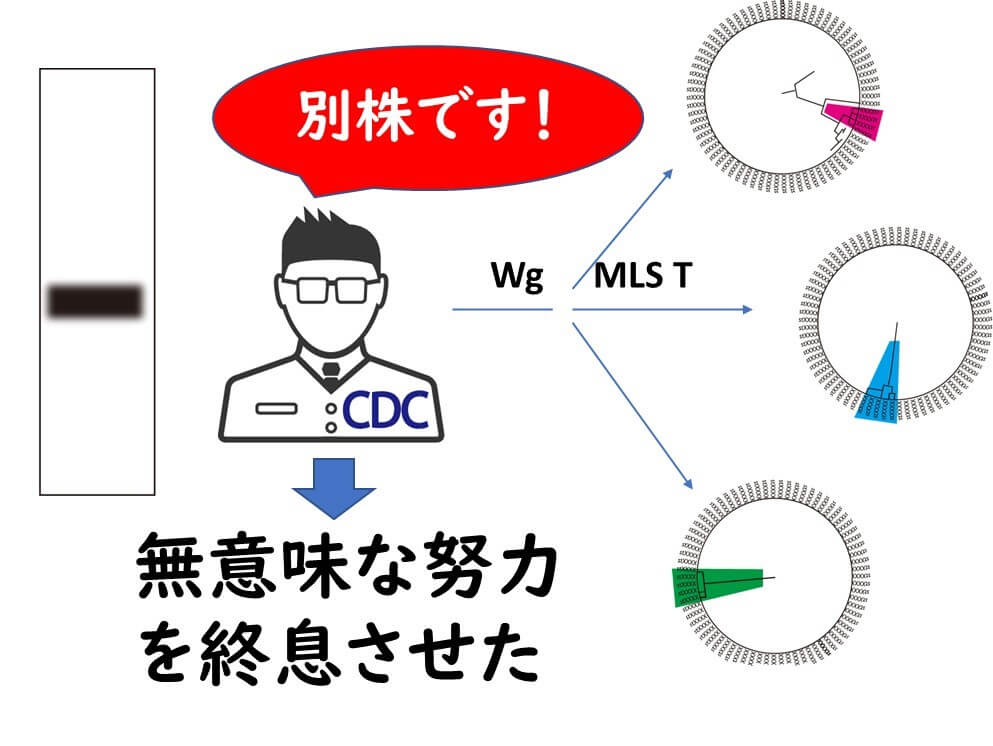
2013年10月に複数州にまたがるリステリア症が報告されました。パルスフィールド電気泳動で分離株は全て同一のバンドを示しました。そのため同一食品を原因とするアウトブレークが疑われました。多くの聞き込み調査が行われたが原因を究明することはできませんでした。ところが2015年1月2whole genome ml ST解析した結果、これらは別の菌株であることが判明しました。
このようにして無駄な疫学的調査が終了させることが出来ました。
Genome Trakrプロジェクトの成功事例パターン④
食中毒の原因究明において、患者が1人である、いわゆる ”散発事例”の原因究明は極めて困難である。患者の自宅で見つかった食品から食中毒菌が分離されない限り、原因究明はほとんどできないのが実態です。
Geome Trakrプロジェクトでは、このような散発事例も解決にも貢献できることがわかりました。
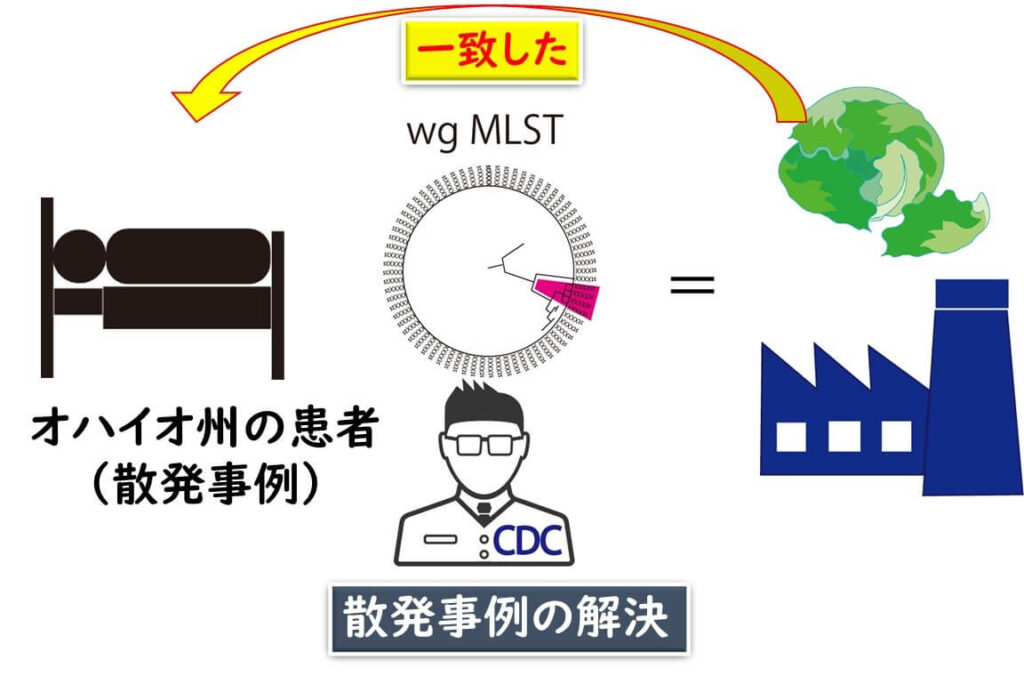
2014年、レタス工場レタスからリステリアが分離され、リコールとなりました。このレタスを原因とする食中毒が起きているか否かについては、不明でした。
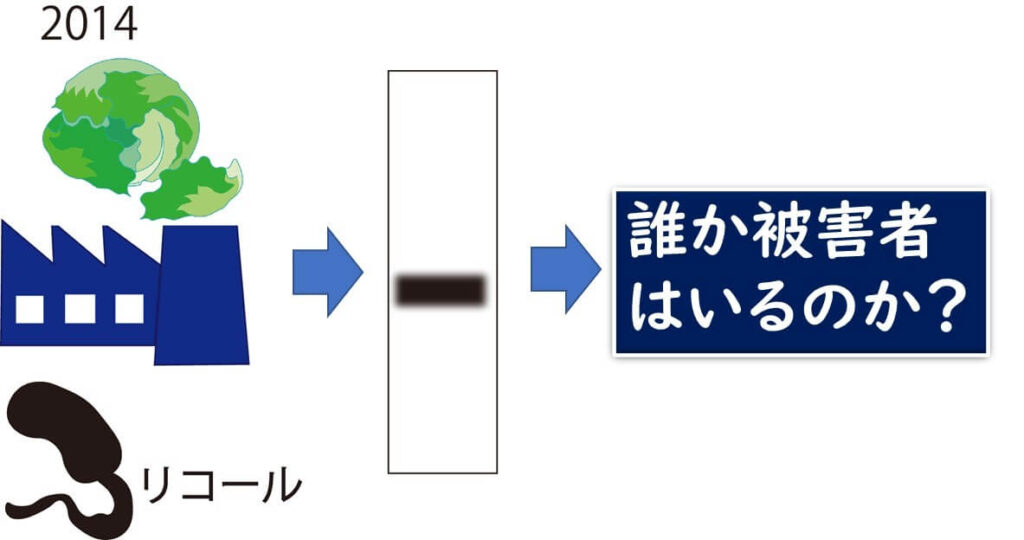
whole genome MLS Tのデータベースからオハイオ州での散発事例での患者分離株と解脱分離株は近似株であることが判明した(≤5 hqSNPs, ≤4alleles)。患者の聞き取りを行ったところ、この工場のレタス食事履歴が判明しました。
次世代シークエンサーを応用した食中毒原因細菌の解析へ、EU諸国でも急速に移行中
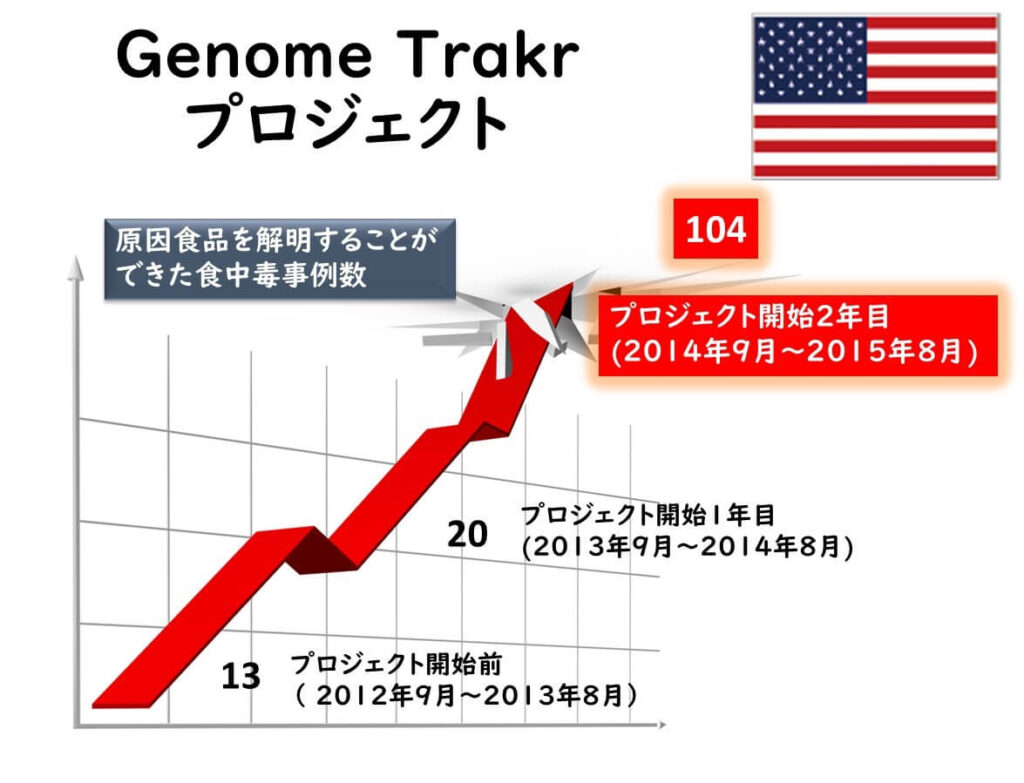
以上のようにリステリア菌についてみると、プロジェクト開始前の1年間における原因食品の究明された中毒件数は13件でした。プロジェクト開始1年目では20件になりました。そしてプロジェクト2年目では、104件にまで数字が跳ね上がりました。
なお、最近では、2021年12月にも、定期検査でリステリア菌が検出されたたためリコールとなったミックスサラダが、過去数年前の未解決リステリア食中毒事例と遡及的に結びつけられたという報告が米国FDAとCDCからありました。しかも、2社の製品が、それぞれ、別の過去の未解決食中毒事件クラスターとそれぞれ、遡及的にリンクづけられています。 次世代シークエンサーを応用した食中毒原因細菌の解析の恐るべき威力を物語っています。
※この報告については別記事で詳しく説明しましたのだ下記記事をご覧ください。
リコールされたサラダのリステリア菌株は次世代シークエンサー解析により過去の原因不明食中毒事件とリンクされた(米国)
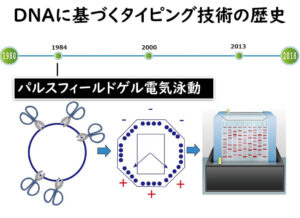
現時点で、日本の 食中毒原因細菌の解析 はパルスフィールドゲル電気泳動法や、また、腸管出血性大腸菌ではMLVA法で行われています。
※ パルスフィールドゲル電気泳動法やMLVA法 については、下記記事にわかりやすくまとめたので、ご覧ください
病原菌や食中菌の感染ルート把握のための分子疫学解析手法(菌株の識別法)のすべてをわかりやすく解説します
しかし、現在、米国に続き、過去数年間で英国、ドイツ、フランスなど、 EU 諸国も続々と次世代シークエンサーによる食中毒の感染経路解析への移行が次々と進んでいます。日本と同様に、これまでMLVA法で食中毒原因細菌をおこなっていたベルギーでも、2021年10月に連邦政府機関(FASFC)の科学委員会が、近い将来、食品の低検査分離株をWGSへ移行開始するようアドバイスしました。現在、ベルギーでは、全国的なWGSの導入はまだですが、一部のラボではこの技術を使用開始し始めています。ただし、利用可能なWGSデータはまだ限られており、一元化されたデータベースはまだありません。
日本でも近い将来、米国の Genometrkrプロジェク のような全ゲノム解析システムに移行することを期待したいと思います。
この論文は2016年に出版され、これまでに194回引用されています(2021年12月scopus調べ)。
Implementation of Nationwide Real-time Whole-genome Sequencing to Enhance Listeriosis Outbreak Detection and Investigation
Clinical Infectious Diseases, 63(3):380–6(2016)
この論文はPubMed Central(PMC)で無料公開されています。