本記事では、微生物同定を遺伝子検査によって行う方法を分かりやすく解説する。細菌の種をどのように定義するのか、OTU(operational taxonomic units)とはなにか、16S rRNAとはなにか、 16S rDNA とは何か、ハウスキーピング遺伝子とは何か、なぜ 16S rDNA 遺伝子解析(シーケンス)が同定に適しているのか、系統解析との関連、種の同定に適している配列領域とはなにか、その検査のメリットと限界、留意点について、解説する。
食品の微生物検査において遺伝子検査を用いる目的
食品の微生物検査において遺伝子検査を用いる目的は大別すると次の二つになる。
- 分離した細菌の種の同定
- 食品からの目的菌の直接検出
本記事ではまず分離した細菌の種の同定に遺伝子検査を用いる方法について説明する。

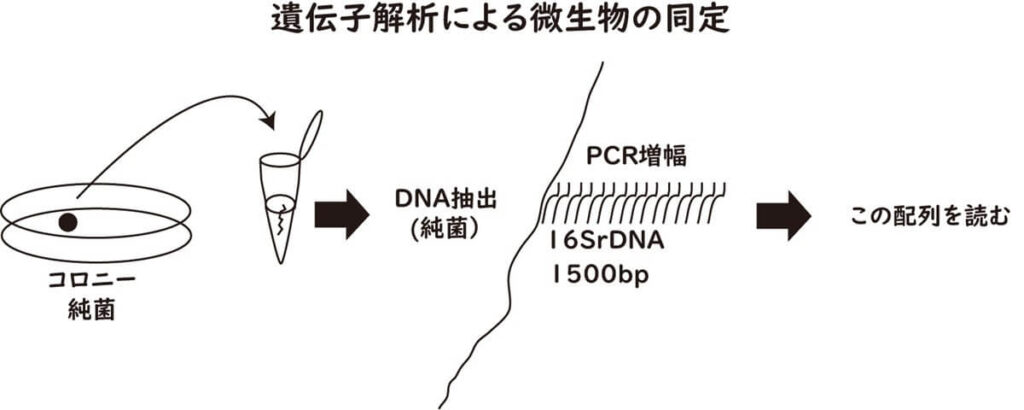
すべての細菌が持っている共通遺伝子の配列を読み、種を同定する
細菌は一般的にはだいたい千個~2千個ぐらいの遺伝子を持ってる。これらの遺伝子は細菌の種類の違いによって様々である。ただし細菌の種を同定するためには、全ての細菌が共通に持っている遺伝子を使う必要がある。例えば大腸菌は乳糖を分解する酵素を持っている。これは大腸菌が属する大腸菌群のグループの特徴である。従って乳糖分解酵素の遺伝子は大腸菌群の中のさらに細かな種を決めていくのには有効である。しかしサルモネラはそもそもこの酵素を持っていない。したがって、サルモネラの種の同定にはこの遺伝子は使えない。
種の同定に適しているハウスキーピング遺伝子 とは?
それでは、全ての細菌が持っている遺伝子とは何か?これらの遺伝子を一般的にはハウスキーピング遺伝子と呼ぶ。ハウスキーピングとは日本語では家事である。全ての人々は色々な趣味や仕事を持っているが、共通な事項として家事を疎かにしてしまえば生活が成り立たない。つまり細菌全体の生命の維持するために必須な根幹的な遺伝子である。このように全ての細菌が持っている遺伝子は、全ての人々が共通して不可欠に行わなくてはならない家事に似ているので、ハウスキーピング遺伝子と呼ぶ。
細菌のタイピングに用いる遺伝子を大別すると、 ハウスキーピング遺伝子 とその他の変化しやすい遺伝子に2分できる。 ハウスキーピング遺伝子 とは、細菌の生存の根幹にかかわる遺伝子であり、たとえばリボゾームの遺伝子(16S rDNA)やDNAの複製に関与するジャイレース遺伝子(gyrB)、ヒートショックタンパクなどのストレスタンパク遺伝子などがこれにあたる。 ハウスキーピング遺伝子 の変異は細胞にとって根幹的な作用に影響を与える。したがって、ほとんどの場合、変異を起こした細胞は死滅するか、すくなくとも、集団の中で淘汰されてしまう。このため、遺伝子の進化速度は遅い。また、これらの遺伝子の変異が子孫に残るか否かについては、細菌のおかれた環境要因の影響はほとんど受けない。いわば、これらの ハウスキーピング遺伝子 の進化は、「進化の体内時計」として捉えることができる。
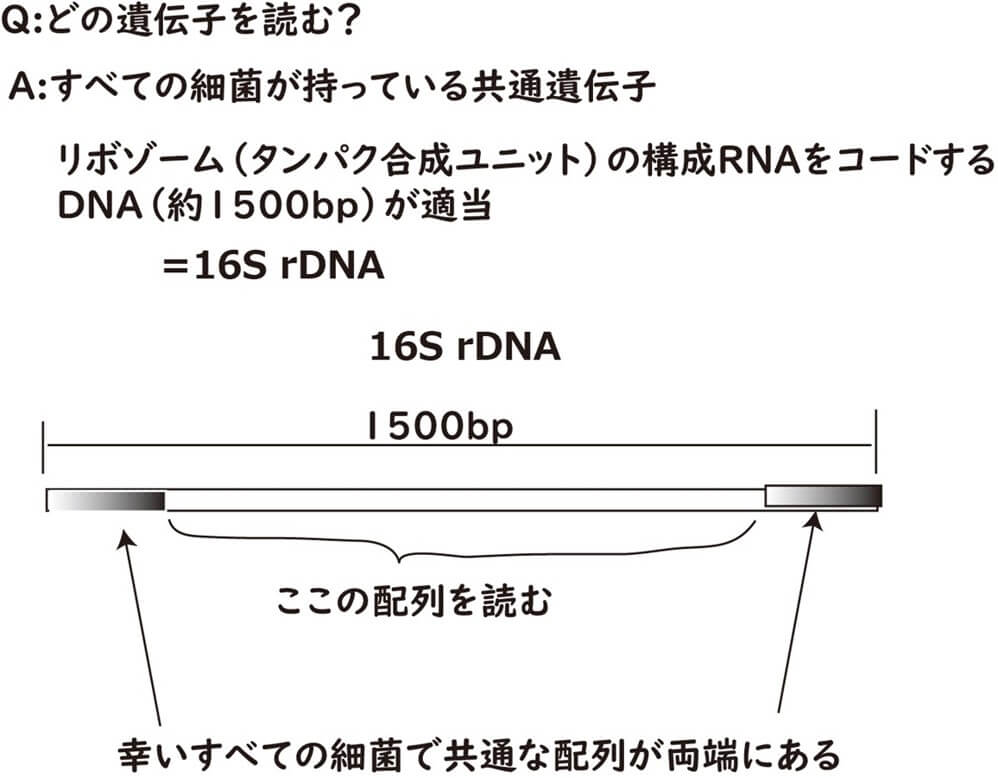
ハウスキーピング遺伝子 はいくつかあるが、代表的なものがリボゾームに含まれている RNA に関する遺伝子(DNA)である。リボゾームは全ての生物がタンパク質を合成するための基本的な細胞内小器官である。したがって全ての生物が持っている。
以上の理由から、細菌の種の同定のためには16 S リボソームに含まれている RNA の遺伝子(DNA)を用いる。すなわち、16SリボゾーマルDNA(16S rDNA)である。
種の同定に適さない遺伝子
一方、細菌は、 ハウスキーピング遺伝 以外に、細胞の生存の根幹にかかわらない遺伝子も多くもつ。例えば、ある糖を代謝する遺伝子とか、鞭毛の遺伝子などはこれにあたる。病原細菌の場合、血清型を決める遺伝子や病原性遺伝子そのものも、菌自体の生存の根幹にかかわらないので(偏性病原体の場合は別)、こちらのグループに分類できる。このような、遺伝子群は、環境の変化に応じて比較的変化しやすい。つまり、変異が致命的なマイナスでない場合は、長い進化の過程でこれらの遺伝子の変異は、 ハウスキーピング遺伝 に比べると子孫に伝えられやすい。このような変化しやすい遺伝子は、鞭毛の有無や病原性、血清型、糖の発酵性など、それを検査する人間から見ると一見して大変大きな差異を生じる場合が多いので、分子生物学的な系統では極めて近縁であっても、表現形質からみると、全くの別種に分類されてしまっていたケースも多い。これらの株を ハウスキーピング遺伝 により本来の進化の歴史時間から細菌の系統を解析してみると、しばしばひとつの種内のきわめて近縁な株であることがあきらかとなっている。
これらの株は、進化時間的に言えば、ごく最近に、変わりやすい遺伝子の変異がおき、環境の変化により選択淘汰され、わたしたち人間からみると随分違う菌のようなったものと考えられる。例えば、Photobacterium damselae(日和見感染菌)とPasteurella piscicida(魚病菌)などは、鞭毛の有無やさまざまな生化学性状の違いから別種として長く扱われてきたが、16S rDNA解析により同一種に統合された。また、大腸菌と赤痢菌などは、リジン脱炭素酵素の有無などの表現形質に差異はあるが、分子系統的には同種といってよい。

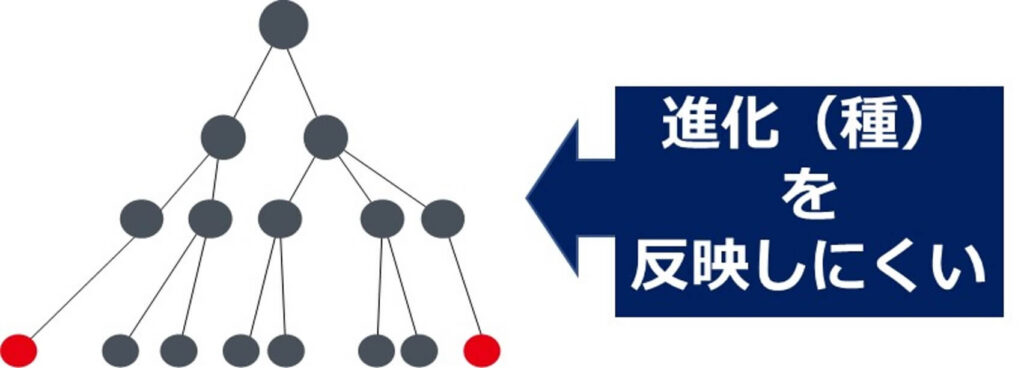
つまり、細菌の種の同定にハウスキーピング遺伝子を用いる理由には、単に全ての微生物がこれらの遺伝子を持っているということだけではない。もう一つの理由がある。それはこれらの遺伝子の中の変異が微生物の進化速度を正確に反映しているという点である。これらの遺伝子のことをいわば進化の体内時計と呼ぶことができる。たとえば、乳糖を分解する酵素は進化の体内時計としては不適当である。
例えば、大腸菌が乳糖がない環境に放出された場合に、乳糖分解酵素を欠落する可能性がある。逆に今まで乳糖分解酵素がなかった細菌にプラスミドなどを通じてその酵素遺伝子が入ってくる可能性もあるからである。つまり、乳糖分解酵素の遺伝子は環境要因によって大きく影響を受ける。このような遺伝子を用いて微生物の進化を理解しようとすると、間違った系統樹を書く可能性が高い。
種の同定に適するハウスキーピング遺伝子
これに対してリボソーム RNA 遺伝子のようなハウスキーピング遺伝子は、少しでも変異が起きてしまうとその細胞は生きていけない。仮に変異が起きた場合にはその細胞が個体レベルで 死んでしまう。その結果、ハウスキーピング遺伝子は自分の子孫にその変異を伝えにくい。仮に進化の過程で変異が子孫に残されたとしても、進化の過程ではとてもゆっくりとした変化になる。従って、細菌の16 S リボソーム RNA のDNAの違いは33億年のバクテリアの長い進化の中で徐々に蓄積された変異だとみなすことができまる。
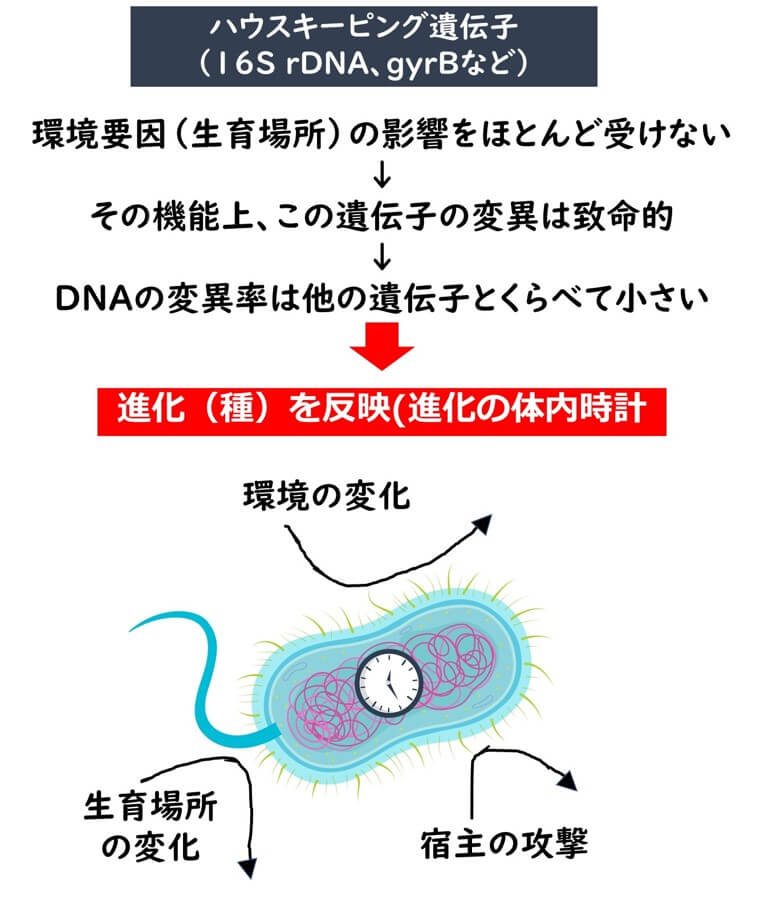
上に説明したように、ハウスキーピング遺伝子の遺伝子の変異が起きたら基本的には子孫を残せない。しかし、後述するように、ハウスキーピング遺伝子であっても遺伝子内の領域によっては変異が起きた場合にそれほど致命的な影響を受けない領域もある。このような箇所の変異が長い進化の歴史上で蓄積されていくわけである。
ただいずれにしても上述したような環境因子によって影響を受けるさまざまな表面的な遺伝子に比べれば、その進化速度は極めて遅い。だから進化の体内時計としてハウスキーピング遺伝子は適していると考えられているわけである。
つまり、ハウスキーピング遺伝子内の変異の度合いが微生物の長い進化の中での系統を反映していると判断してよいことになる。
以上、細菌の系統樹を書く場合に ハウスキーピング遺伝子 を用いると、系統を正しく反映するが、その他の遺伝子を用いると系統を反映しない場合がある。したがって、細菌の系統を正しく反映させたい場合、言い換えると、進化的時間通りに系統樹を書きたい場合は、 ハウスキーピング遺伝子 を用いる必要がある。
なぜ、リボゾームにRNAがあるのか?
ところでなぜリボソームに RNA が存在するのだろうか?
リボソームは細胞内のタンパク合成装置である。タンパク質でできた細胞内小器官である。 DNAを 鋳型にコピーされたメッセンジャー RNA がリボソームに運ばれて、リボソームに結合する。この際、メッセンジャー RNA がリボソームタンパクと結合するためには、リボソームタンパク側にも RNA が存在しておく必要がある。すなわち、核酸塩基の相補性を利用してメッセンジャー RNA をリボソームタンパクにつなぎとめるわけだ。
リボソームを 列車の駅で例えるなら、メッセンジャー RNA は次々と駅の構内に入ってくる列車である。そしてその列車が正確にホームの停止時に泊まるように、駅の構内に敷かれた線路がリボゾーマルRNA ということになる。もちろんこの RNA も元々は DNAの暗号情報によって作られリボソームに送られたものである。従って微生物の同定には、このリボソームの RNA をコードする元々の DNA の配列を用いるということになる。すなわち、16S rDNAである。
なお、16 S の意味についても説明しておく。まず、「S」はタンパク質を超遠心分離機で沈降させる際の速度を表す係数である。すなわち、タンパク質の大きさを表す単位と考えれば良い。細菌のリボゾームは70 S のリボソームを持ち、さらにこれが50 S と30 S のサブユニットに分かれる。16 S リボソーム RNA は30 S のサブユニットに結合している RNA である。
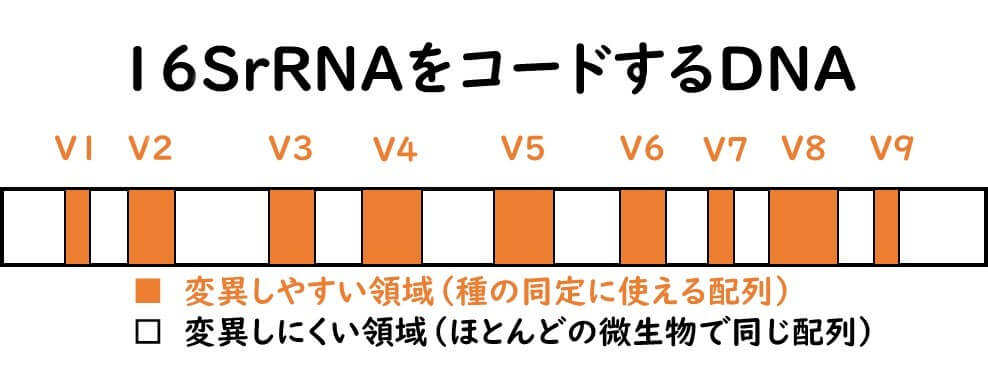
16SrRNAの立体構造で変異しやすい場所
16 S r RNA は約1500の塩基よりなる。下の図に示すような立体構造をとる。下図のように、お互いに塩基の相補的な結合によって幹のような構造をとっている部分と、ループ状に展開している部分がある。
幹のような構造をとっている部分は、 16S rDNA 塩基配列に変異が起きると、それからできた 16S rRNA 全体の立体構造に影響を及ぼす。
一方ループ状のところに変異が起きても、この部分は塩基の相補的結合によって結合しているわけではなく、単鎖として配置されている部分である。 従ってここの部分の塩基配列が変異が起きても、16 S リボソーム RNA 全体の立体構造の大きな変化は起きない。
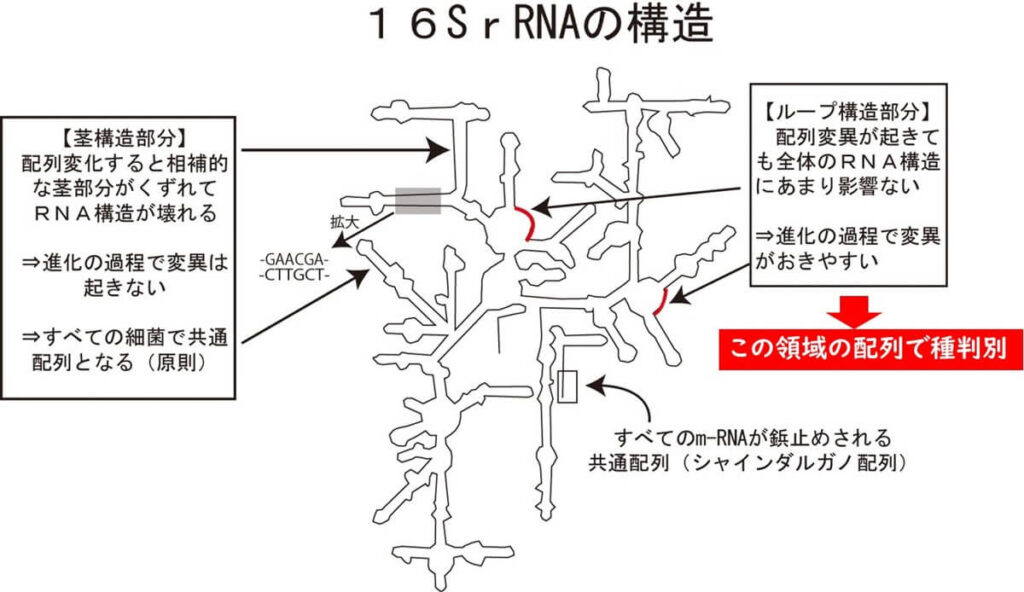
以上の理由により、 16S rRNA の全体の配列で、幹の部分に関しては変異が起きにくい。変異が起きても個体細胞レベルで死滅してしまい、進化の上で残らない。一方、ループの部分は、変異が起きても、リボソーム RNA としての機能を保持しやすく、したがってその変異は子孫へ残りやすい。この変異の蓄積こそが細菌の系統進化ということになる。この部分は選択圧とは無関係に自然発生的に蓄積していくので進化の上では体内時計として見ることができるわけだ。この配列で種の同定を行う。
また、16 S リボソーム RNA の1500塩基全体で種の同定をするのが、実験的に煩雑な場合には、ループ状の可変領域のみで便宜的に種の同定を行うことも広く行われている。例えば前半の500bPを読んだり、あるいは、V3-V4領域だけを読むことによって便宜的に種を推定することも行われている。
16S rDNA 同定は OTU(operational taxonomic unit) に過ぎない
食品から特定の微生物を検出する多くの場合、「種」を判定する。そこでまず、遺伝子配列と種との関連性はどこまで信用できるかということについて述べておきたいと思う。
細菌の「種」は高等植物と異なり形態学的な特徴が信頼できず、「種」に関する完全な統一的な見解は存在していない。
そもそも、細菌のように短時間に1個体が2個体に分裂を繰り返していく生物においては、遺伝子の変異がおきやすい。従って、遺伝子配列がわずかに異なるクローン集団が一つの基準株の周囲に存在している。このようなクローン集団の中で、「種」の線引きをすることは容易な作業ではない。
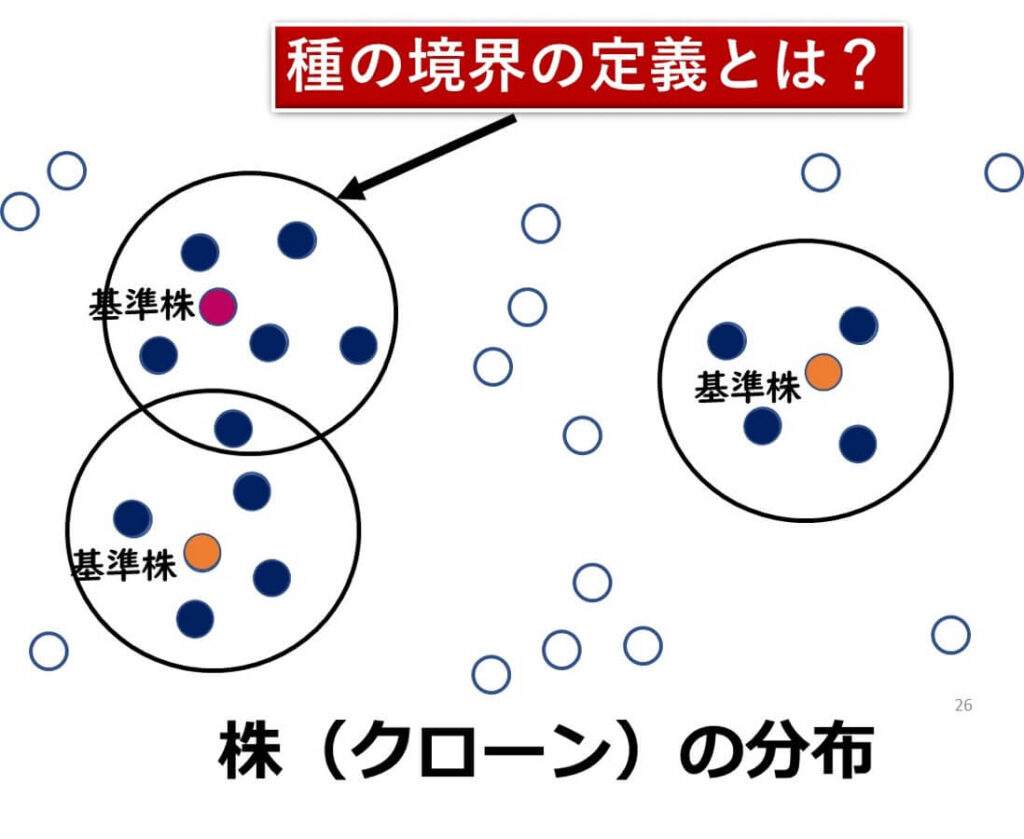
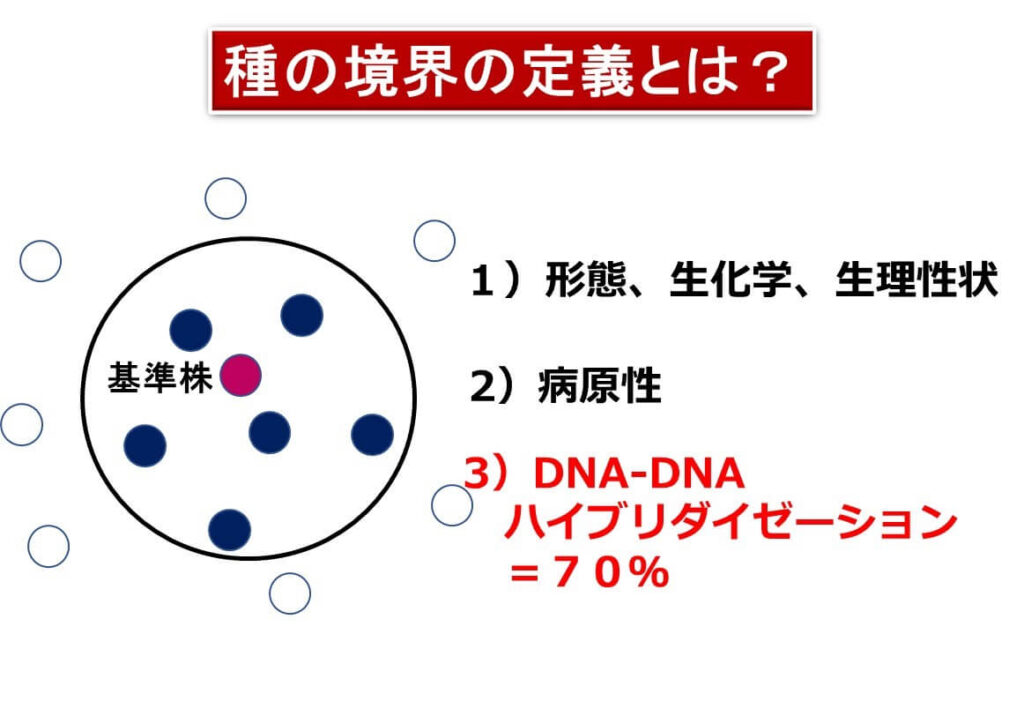
現在の細菌学においては、グラム染色、形態、生理的な正常や生化学的な性状などの総合的な表現形質おもとに種が定義されている。
また、1990年以降、細菌のゲノムの科学が発達し、細菌株同志のDNAの相同性、すなわちDNA-DNA交雑形成率が基準とし用いられるようになった。現在の分類学では、 バクテリアは基準株 (type strain) とのDNA-DNAの相同性が70%以上の場合に同種であると定義される。少なくともこれが遺伝情報による種の同定の基本である。
では16 S ribosomal DNAによる種の同定とはいったい何であろうか?
ウーズ博士が微生物の系統解析に16 S rDNA を用いたのは1977年のことである。それ以来16 S ribosomal DNA解析は微生物の分類や生態学において欠かせないツールとなった。
DNA-DNA交雑形成試験は非常に操作が面倒くさく、正確なデータを出すには高度な技術を要する。筆者も1990年代初頭にこの技術を試みて実験を行ったことがある。しかし、習得するだけでも一ヶ月以上を要し、またその結果についても技術レベルとの関係でどこまで信用できるのかと自信ももてなかった。ちょうどその時期に、16 S rDNA による同定が登場してきたわけだ。
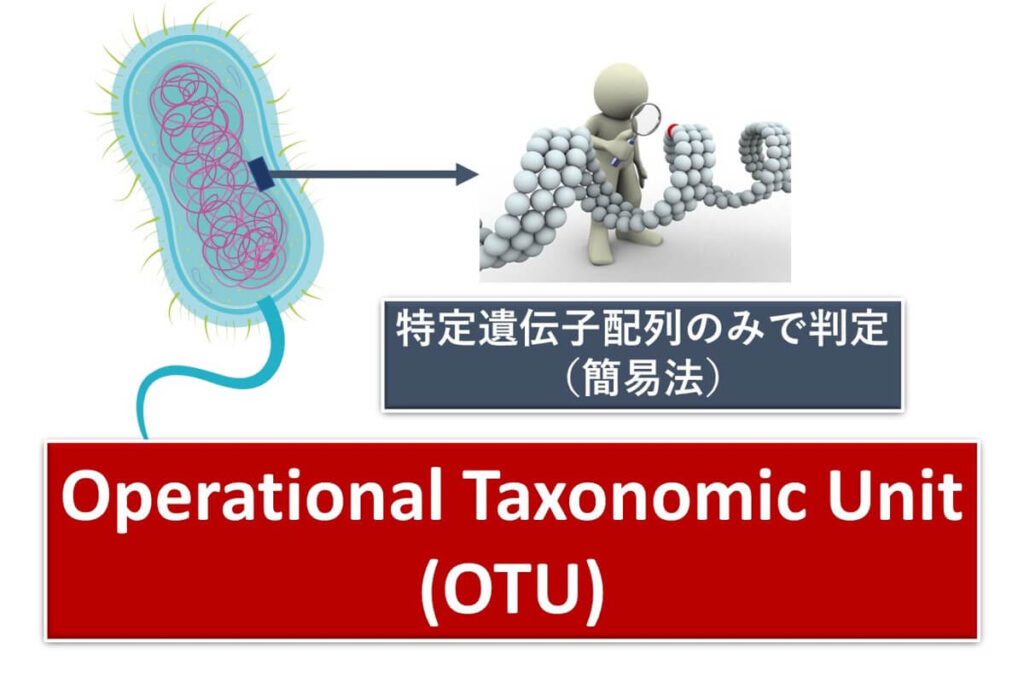
ここで是非理解しておかなくてはならないのは、 OTU(operational taxonomic unit)という言葉だ。OTUとは、細菌の特定の塩基配列をコンピュータ上でその類似度を指標に分類したときに得られる単位をいう。ある一定以上の類似度の閾値をもって一つのグループが構成される。このように、ひとつのこのような配列の類似度だけから種を判定しようとする分類上のユニットがOTUである。すなわち、 16 S rDNA を用いて判定される種は正確には種ではなく、OTUである。 16 S rDNA 全長解析で得られるOTUを便宜的に種として 扱ってきたにすぎない。
16 S rDNA 同定は100%信頼できるものではない
16 S rDNA が世界に登場した1990年代以降2000年半ば頃までは、分類学のコンセンサスとしては、 16S rDNA の全長1500塩基を解析した場合、相同性が97%以上であれば同種である可能性が高いと整理されてきた。これは、相同性が97.0%未満の場合、DNA-DNA相同性は70%以上を⽰さない場合が多いことによる。
しかし、 細菌種によっては DNA DNA ハイブリダイゼーション70パーセント以上の定義と比較すると16 S ribosomal DNA の 97%の閾値は低すぎるというケースも多い。最近では、相同性が98.7%以上を持ってきて同種とみなすべきだとの考え方になっている(下記参考文献参照)。
Stackebrandt,E. and Ebers,J.
Taxonomic parameters revisited: tarnished gold standards. Microbiol. Today, 33, 152–155(2006)
さらに2018年に報告されたレポートでは、16 S 全長解析での種の定義として相同性を97%から99%に変更すべきだという提案もなされている (下記参考文献参照) 。
Edgar RC (2018).
Updating the 97 % identity threshold for 16S ribosomal RNA OTUs. Bioinformatics. 15;34(14):2371-2375(2018)
Open access
比較的多くあるパターンとして16 S リボソーム DNA の相同性が98%であるにも関わらず、 DNA DNA ハイブリダイゼーションを行うと70%よりもはるかに低いケースである。すなわち16 S リボソーム RNA では特定の特定の種と推測しても、 実際にはその種とはかけ離れた主である可能性があるということである。なお、下のイメージ図に示すように、 16 S リボソーム DNA の相同性が98%以下であるにも関わらず DNA DNA ハイブリダイゼーションの値が70%以上になるというケースはほとんどない。従って16 S リボソーム DNA の相同性が98%以下では同種になる可能性はほとんどないという判断はできる。問題はその逆で、 16 S リボソーム DNA の相同性が98%だから必ず同種になるかというと、必ずしもそうではない。 16 S rDNA 同定 では、このような誤同定の可能性を念頭に入れておかなくてはならない。
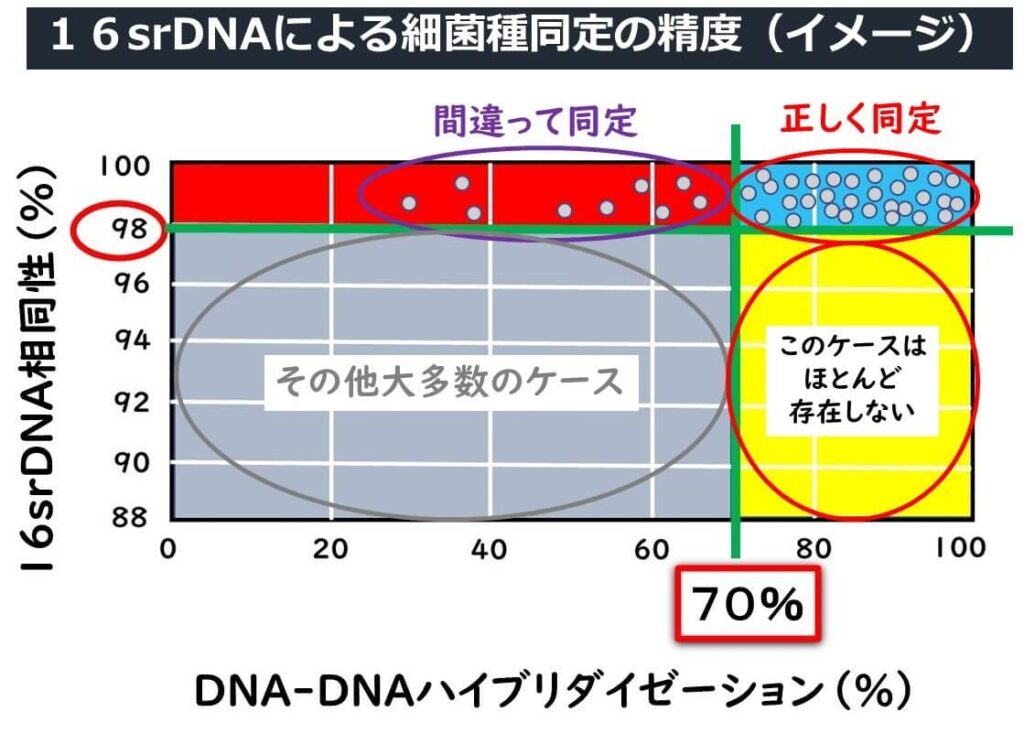
また、そもそも、 16 S rDNA による種の判定そのものに欠陥がある場合もある。細菌によっては、16 S ribosomal DNA で同種判定したOTUの中に球菌と桿菌が混在していたり、さらにはグラム陰 性菌と陽性菌が混在している場合などもある。また酸素要求性などの根本的な生理性状が根本的に異なる場合などもある。
すなわち16 S ribosomal DNA の全長解析による細菌の同定法そのものに種の同定ツールとしていくつかの欠点もあることをまずは理解しておくべきである。

このように、 16 S rDNA による種の同定は、正確には 上述したoperational taxonomic unitを判断しているだけであることを常に念頭に入れておく必要がある。細菌学における「同定」を最終的に確定するためには、Bergey's Manual of Systematic Bacteriology による各種の表現型形質や DNA DNA ハイブリダイゼーションを行う必要があるということを注意しておく必要がある。
もちろん食品企業においての日常的な検査における細菌の同定に、Bergey's Manual of Systematic Bacteriology は必要はない。 16 S rDNA による種の推測だけでも実用的にはほぼ事が足りる場合が多いだろう。このような 16 S rDNA による種の推測の限界については常に念頭に入れておく必要があるということをここでは述べた。