次世代シーケンサーを用いることにより、ハイスピード、低コストで、DNAの配列を読むことができるようになった。食品から直接多様な微生物のゲノムを抽出する16SrRNAアンプリコンシーケンスにより、培養法に比べると格段のスピードで細菌叢を解析できることが可能となった。本記事では、 16SrRNAアンプリコンシーケンス解析 が食品産業界のどのような場面で活用できるのかについて、未来を見据えた展望をしてみたい。
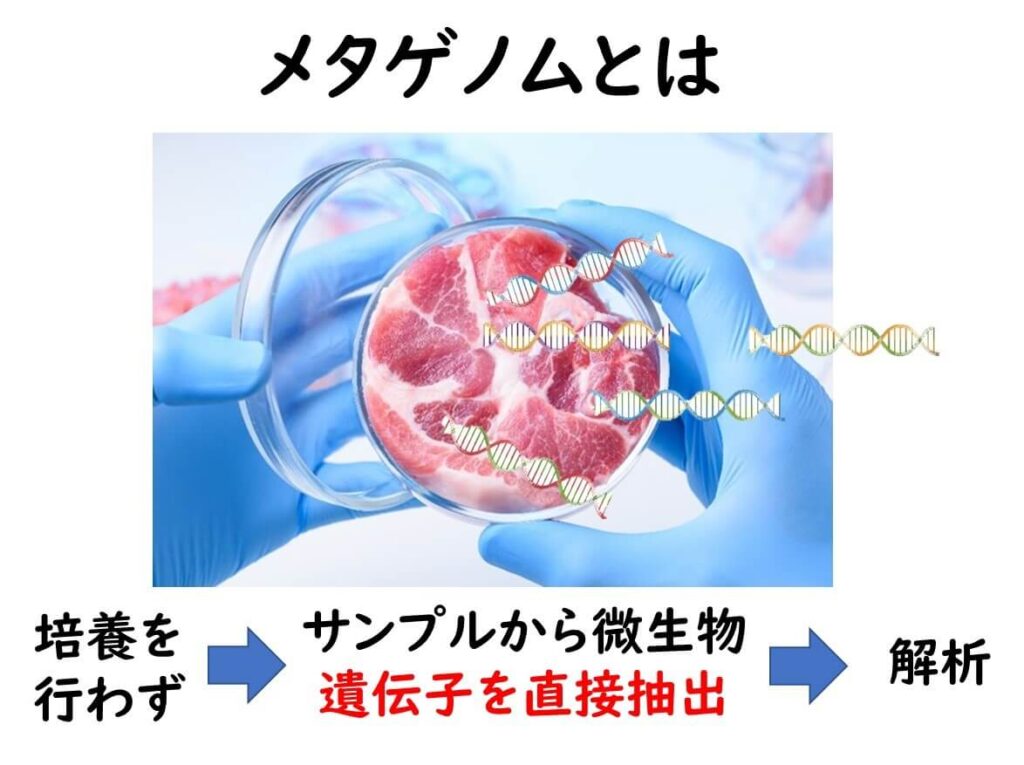
メタゲノム解析とは?
メタゲノム解析の「メタ」(meta-)の意味は「超越」という意味である。直訳すればゲノムを超越しているという意味になる。微生物学分野においては、培養を省略し、そのままサンプルからゲノムを抽出し、これらを全て網羅的に解析する方法のことをメタゲノム解析と呼んでいる。
16SrRNAアンプリコンシーケンス はメタゲノム解析ではない
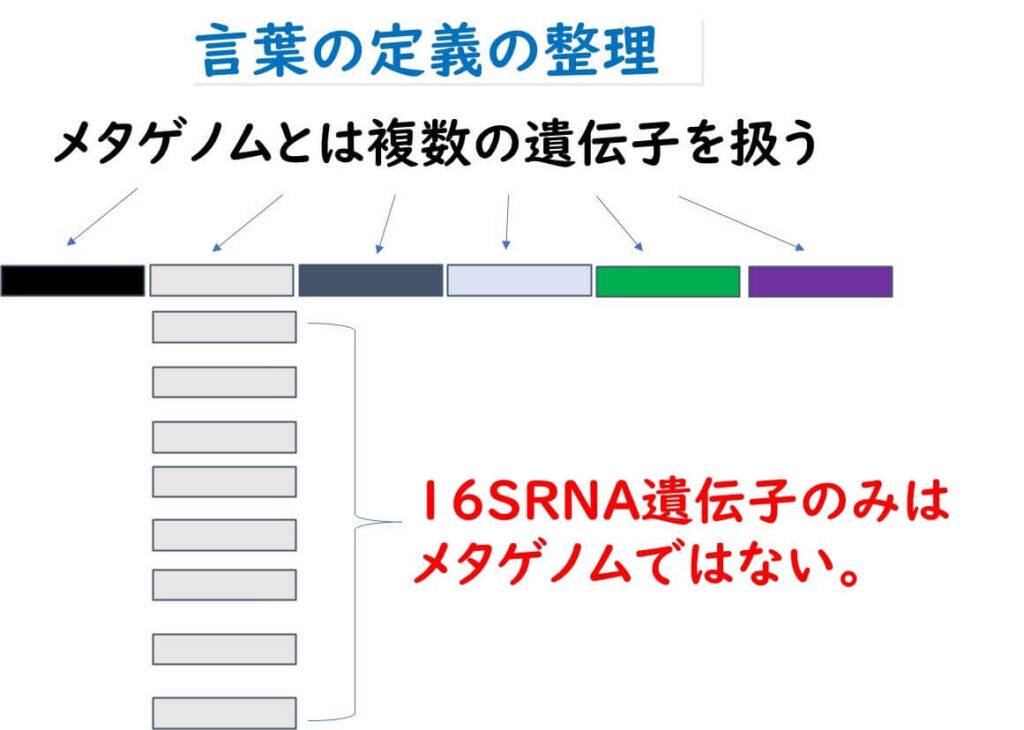
これまで、環境や食品中から直接雑多な細菌群集の遺伝子を抽出して、16 S リボソームRNAの遺伝子のみを用いて微生物群集を調べる方法については、メタゲノムという言葉を使う場合が多かった。事実、過去10年間に出版された論文においてもメタゲノムという言葉が使われている場合の頻度が高い。またホームページなどでも、この手法についてメタゲノムという言葉を使っている場合がある。

しかし最近の微生物ゲノム専門家の間の議論として、16 S リボソーム RNA の遺伝子を増幅して微生物群集構造を調べる方法についてメタゲノムと呼ぶべきではないという見解が出されている(下記サイト参照)。
16s rDNA targeted NGS is Metagenomics or Metabarcoding?

なぜならばこの手法は16 S リボソーム RNAをコードする DNAという 単一遺伝子のみを取り扱っているからだ。メタゲノムと言う言葉はその定義上複数の遺伝子を取り扱う手法についてのみ該当する用語である。 もちろん現在でも専門家の中には、環境や食品などの多様な遺伝子を含んだサンプルを一斉に抽出して解析するのであるので、この部分でメタゲノムと呼んでも良いのではないかという意見も存在する。しかし遺伝学のコアな専門家の間では、単に環境や食品などから多様な遺伝子をサンプルとして抽出した場合をメタゲノムと定義するのは間違いであるとの見方の方が 優勢だ。サンプルレベルでメタサンプルであってもメタゲノムという定義にはならないとの見方である。
したがって今後正しい言葉としては、メタゲノムではなく、例えば、16SrRNAアンプリコンシーケンス、16SrRNAメタバーコーディング、もしくは16SrDNA解析などの言葉のほうがが望ましいであろう。本ブログでは便宜的に16SrRNAアンプリコンシーケンスに統一することとする。

16SrRNAアンプリコンシーケンス解析の 食品微生物検査への応用
16SrRNAアンプリコンシーケンスによるアプローチは、食品産業にどのような未来をもたらすだろうか。
食品の安全性の面と食品の品質の面とは2つの側面を比較してみよう。
- 食品の安全性面
食品の安全性評価においては現時点ではその活用範囲はあまり大きくないかもしれない。なぜならば食中毒菌をモニタリングにおいては、リアルタイム定量PCRなど、さまざまな優れた検出技術がすでに確立しているからである。ただし16SrRNAアンプリコンシーケンスは、あらかじめ標的微生物が定まっていなくても、手広く検出が可能であるという点において優れている。守備範囲の広さでは16SrRNAアンプリコンシーケンがリアルタイム PCR に対して優位性を持つ。
しかし、 16SrRNAアンプリコンシーケンスで食中毒菌をモニタリングする場合の弱点として、その検出感度が挙げられる。食中毒菌の場合は、25gあたりに1 cfuを検出する必要がある。食品には多数のバックグラウンド菌が存在する。例えばバックグラウンド菌数が105cfu/gだったと仮定してみる。16S-rRNA アンプリコン解析の感度を上げることは可能であるとはいえ、105リードの中から1リードを検出する食べる目的でこの方法を用いるのは賢明ではない。感度を上げるほどコストが加算される。この点では現時点では食中毒菌の食品からの検出についてはPCR法のほうが技術的な優位性がある。したがって、特定の食中毒菌の検出など標的が定まっている安全性検査においては、 16SrRNAアンプリコンシーケンスを使うのは、現時点では賢明な方法とは言えないかもしれない。
- 食品の品質検査。
一方、16SrRNAアンプリコンシーケンスは、現時点では食品の品質検査においての方がを活用しやすいだろう。
すなわち、微生物のフローラ解析が必須な分野である。NGS が登場するまでは、 例えば、筆者等の研究室においては、一つの食品の微生物フローラを解析するのに大学院修士課程の学生が1年かけて解析を行う状態であった。
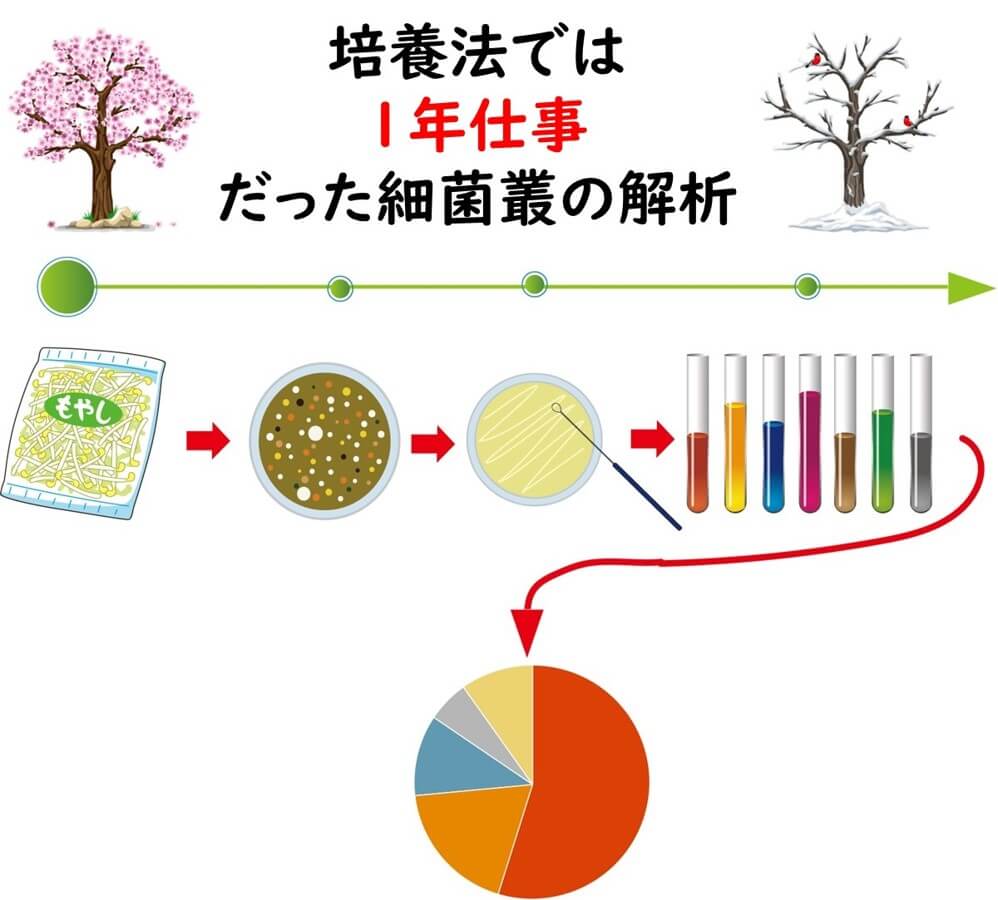
しかしNGS の登場により、同じ作業がわずか1日で行えるようになった。このように微生物フローラの解析が重要となる食品分野においては16 S アンプリコンシーケンスの威力は凄まじいものがある。
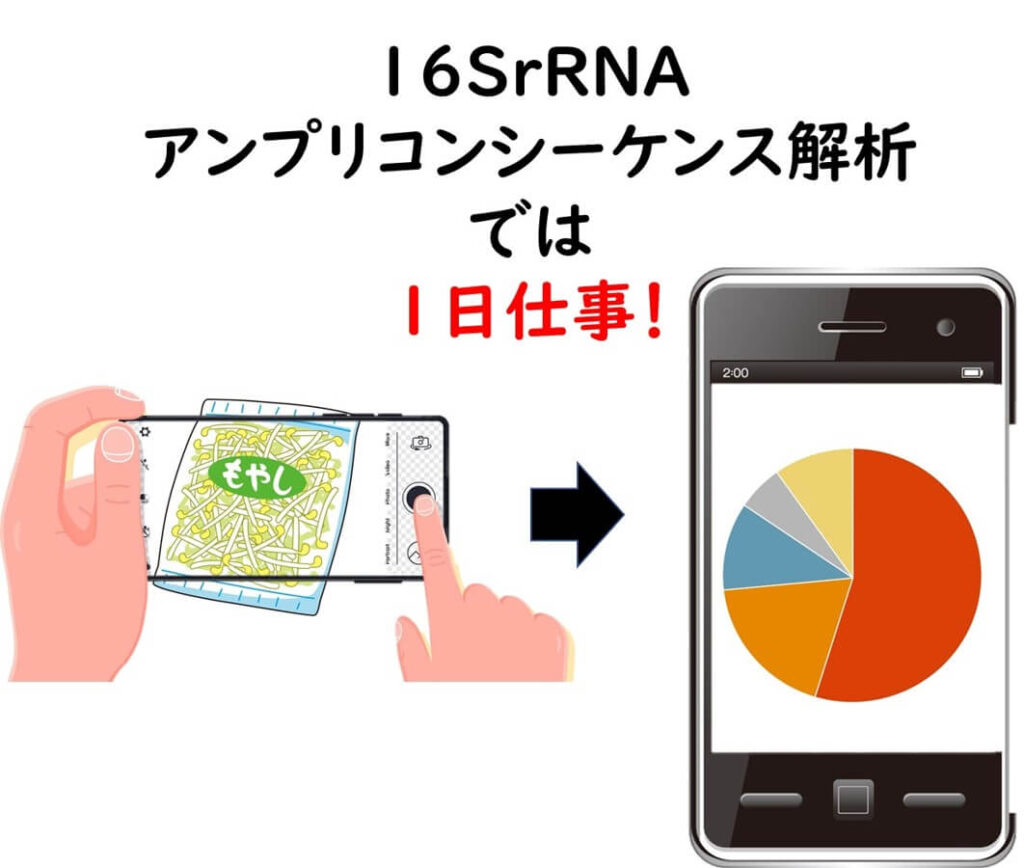
それでは食品産業界においてこの技術はどのように活用ができるであろうか?
食品産業界においての日常検査は大腸菌検査や一般生菌数検査が主体となっている。一般生菌数は、微生物の数によって食品や食品工場ラインの汚染度を測定するものである。あくまでも一般生菌数の総数としての数で判断をしている。これはいわば空港の荷物検査で、カバンの大きさでリスクを判断しているもののだ。

しかし、実際には同じ一般生菌数であっても、その中身、すなわち細菌叢の違いによってその評価は大きく異なるはずだ。空港の手荷物検査で言えば、x線によってカバンの中身を透視するようなものだ。このように食品中の微生物の数を見るだけではなく、その細菌叢を見ることにより、リスクの評価法としては格段と精度が上がる。
どのような技術もそうであるが、その技術が技術的に困難であったり、高価であったりする場合には、その技術の用途については思い浮かばないものである。しかし。その技術が廉価で簡単にできるようになると、その応用性の広さに初めて気がつくものである。次世代シークエンサーにより手軽に可能となった16SrRNAアンプリコンシーケンス技術でも同様のことが言える。

そこで、以下に、この技術をどのように活用できるのかについて以下私見を述べたい。
クレーム対応
自社の製品に原因不明の異常が起きた場合、16SrRNAアンプリコンシーケンスを使うことは有効であろう。例えば消費者からのクレームなどで、ネトや膨張などの異常品が起きた場合、一体どのような菌によってこのような現象が起きてるのかがわからない。このような場合に、NGSの登場の前までは一般生菌数のプレートカウントを行い、代表的なコロニーをピックアップしてそれらを純粋分離してその菌について同定を行なっていた。

しかし、培養法でこのような検査を行う場合の問題点は、培養の対象とするクレーム品の微生物の培養条件がわからないという点である。培養条件を間違えて選択することによって、微生物が何も検出されないこともありうる。
例えば真空包装した食品のチルド温度(10°C)での賞味期限設定実験を行う場合を想定してみよう。自社の製品の賞味期限を設定することが目的なので、法定で定められている一般生菌数の測定条件で行う必要はない。しかし、やはりここは法定で定められてる一般生菌数の方法に従いでデータを出すべきか、あるいは別の方法を取るべきか迷うところである。もちろん、一般生菌数測定法では、嫌気性菌を見ることはできない。
また、一般生菌数におけるPCA培地を用いた場合、この食品中の腐敗の主要細菌が変敗乳酸菌であった場合には正しく測定できない可能性がある。乳酸菌の場合には、 MRS培地などの栄養豊富な培地が必要になる。
このように考えていくと、どのような培養条件でデータを出すべきかについてはそれぞれの食品毎で迷うところである。

また、どのような培養条件で培養をするにしても、微生物培養には時間を要する。
クレーム食品での原因究明には時間が重要な要素となる。特にチルド食品で低温細菌が変敗の原因だと推定し、低温培養を行う場合にその培養時間は数日を要する。さらに。そのあと主要菌叢と判断されるコロニーを純粋分離し。微生物の同定を行う時間も必要となる。
また膨張した包装食品などのクレーム食品では、その製品が持ち込まれた時点で既に膨張原因となった菌がパッケージの中で死滅している可能性もある。このような場合には、いくら培養法で培養を試みても何も微生物が生えてこないということもありうる。

このようにクレーム品の原因究明において培養法に依存してその原因究明を行う方法は、時間を要する可能性や、そもそもの培養に失敗する可能性など、多くの問題をを抱えている。

16SrRNAアンプリコンシーケンスでは、食品から直接 DNA を抽出してそのまま微生物フローラ情報を得ることができる。この場合はその製品の中で、分析時に微生物の生死にかかわらず、その製品中で最も菌数の多い代表的なフローラをまずは知るということが重要である。

これを知ることによって何らかの対策の手がかりを得ることも可能だろう。また、 16SrRNAアンプリコンシーケンスによるこれらのデータを過去に蓄積していれば、これらの菌が汚染した工場のラインの箇所なども場合によってはすぐに思い当たる場合もあるかもしれない。

もちろんこれらの細菌叢が分かったところですぐに問題の解決に結びつくとは限らない。しかし少なくともいくつかの可能性を絞れるかもしれない。 16SrRNAアンプリコンシーケンスは、100%解決に結びつけられる手段としてではなく、さしあたっての無駄な可能性を排除したり、可能性の高い所に絞り込むための道標には使えるだろう。たとえば、見知らぬ街を訪ねて近くにレストランがあるかどうかを検索するのにスマホがあると便利である。近くに全くレストランが存在していないことや、あるいは複数のレストランが存在しているという程度の情報は入ることができる。このように可能性を絞り込むことができるというのが16SrRNAアンプリコンシーケンスの利点である。

さらに 16SrRNAアンプリコンシーケンス のデジタルデータをパソコンの中に蓄えておけば、例え5年前であっても同じような問題が生じた際に、これらのデータベースと参照することによって、迅速に今日の問題を解決することができるかもしれない。
同様に、同じ会社の別べつの工場で起きた問題などについても。微生物叢の比較によって問題解決の道筋を見つけることができるかもしれない。データベース化の利点も、 16SrRNAアンプリコンシーケンス を用いる利点である。

工場のゾーン管理
16SrRNAアンプリコンシーケンスは食品工場の床や ラインなどの洗浄後の細菌叢を日常的にモニタリングすることにも利用できるだろう。洗浄によって除去されにくい微生物叢(工場にしぶとく生き残る細菌群)に関するデータを取っておくことは有効であろう。
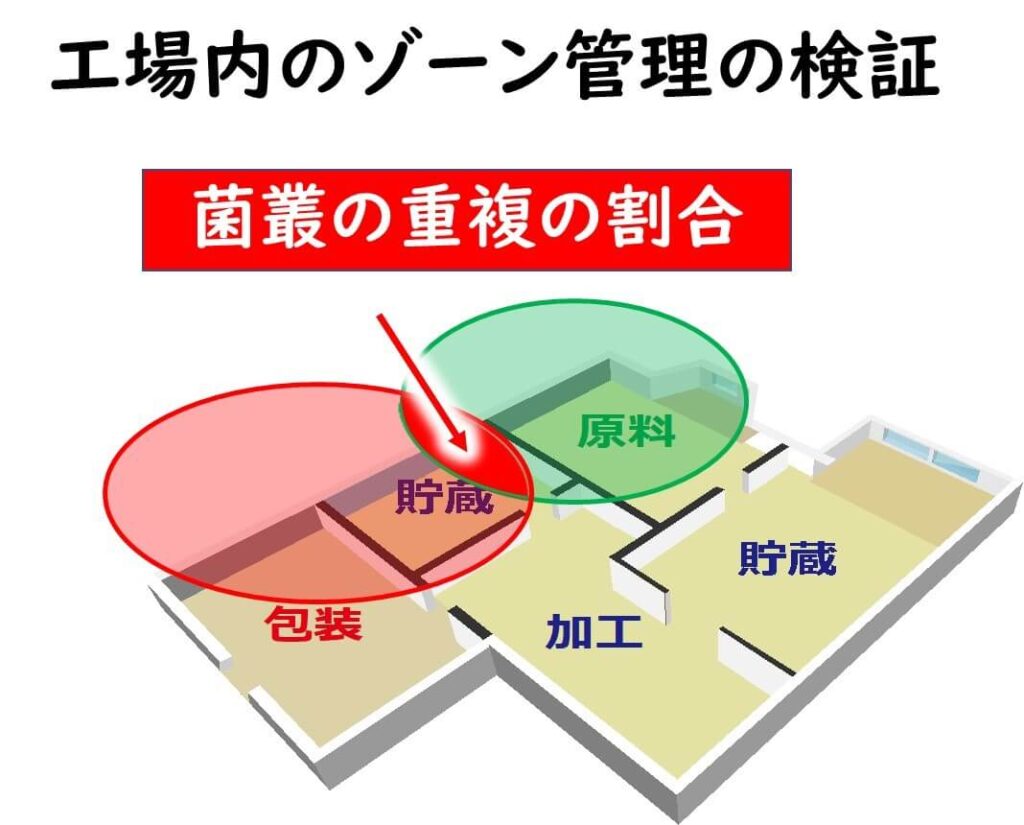
また、食品工場におけるゾーン管理の有効性の検証にも16SrRNAアンプリコンシーケンスを利用することができるだろう。例えば原料エリアとクリーンな包装エリアの16SrRNAアンプリコンシーケンスを行うとする。 2つのエリアが全く隔離されているならば、そこに生じてくるOTUに大きな重なりは生じないだろう。しかしながら二つのエリアに交差汚染が頻繁に起きてるならば、2つのエリアで共通するOTUの存在割合が増えてくることが予想される。極端な話、ふたつのエリアに全く差がないならば、円グラフがほぼ重なり、共通の OTUで占有されることになる。この円グラフの重なり具合、すなわち二つのエリアでの共通のOTUの存在比がエリアのゾーン管理の有効性の定量的指標になり得る可能性はある。
原料の微生物学的品質モニタリング
農作物や魚介類に付着している微生物叢は環境中の微生物叢の影響を強く受ける。NGSの登場前は微生物叢を解析することは時間のかかる作業なので、原材料の産地特定に微生物を利用しよう試みはほとんど行われていなかった。しかしNGSが普及するにつれて、食品中の細菌叢が素早く分かるようになり、このような試みも可能となっていくであろう。現時点ではまだ十分なデータが蓄積しているわけではないが、今後、16SrRNAアンプリコンシーケンスで得られる食品中の微生物相から食品の産地や食品の生産環境についての有益な情報を得ることができる可能性は秘めている。
死菌情報だからこそ役に立つ場合
順不同になるが、下記の留意点の欄で16SrRNAアンプリコンシーケンス の欠点として死菌の DNA を検出してしまう点について述べている。しかし、 16SrRNAアンプリコンシーケンス が死菌も検出してしまうという事実は、必ずしも欠点ばかりとも言えないかもしれない。
たとえば、海外などから仕入れた野菜や肉などの一般生菌数が著しく低い値であったとしよう。 これらの食材の衛生管理状態が悪く、極論すれば腐敗状態になった野菜や肉であっても、次亜塩素酸ナトリウムなどで殺菌処理を行えば、一般生菌数は低い値となる。
※一般生菌数と殺菌処理に関しては下記の記事をご覧ください。
一般生菌数の意味を正しく理解しよう
このような場合に、原料の受け入れ側で16SrRNAアンプリコンシーケンスで細菌のフローラを見ることができれば、微生物学的な品質について情報を得ることができるかもしれない。
新鮮な野菜や魚が新鮮な状態であればその細菌叢はきわめて多様である。生鮮野菜や魚が腐敗をしていくと細菌のフローラは単純化されていく。特に、野菜や肉においては、腐敗が極度に信仰すると、腐敗菌叢特有の細菌種(specific spoilage oraganism、SSO)が出現することが知られている。つまりこのような細菌種は腐敗の指標になり得る。
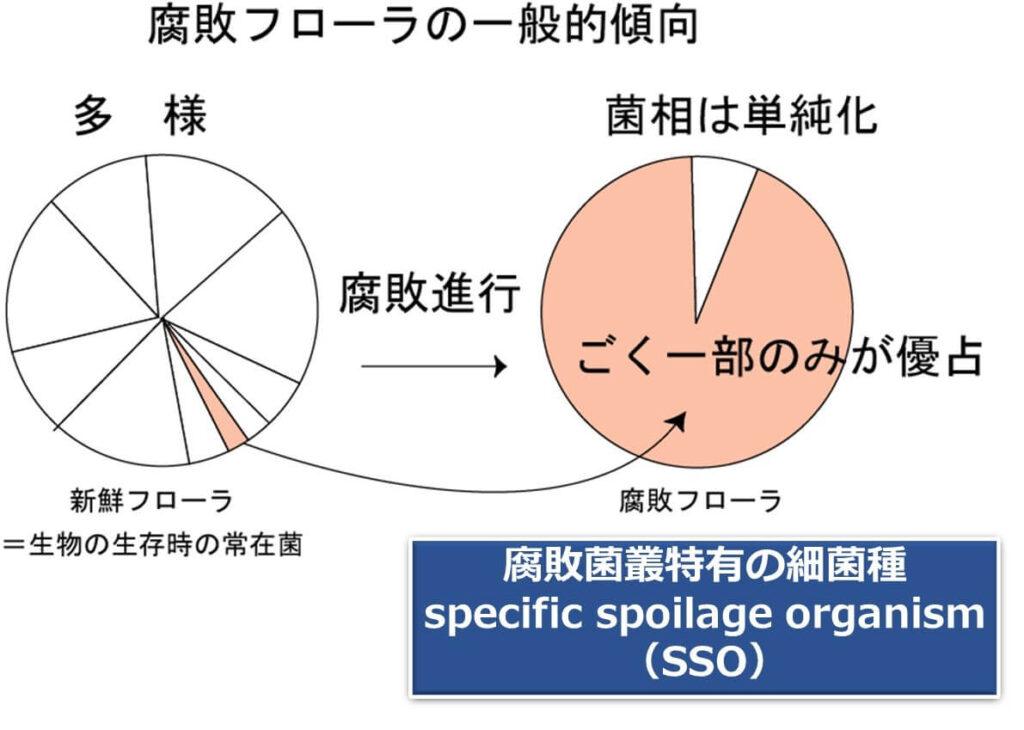
したがって一般的には細菌叢を見るだけで、肉や野菜が新鮮さや腐敗の進行について判断できる。原料を受け入れる企業での16SrRNAアンプリコンシーケンス の結果、単純な細菌叢や、あるいは、腐敗状態で観察されるような細菌叢が観察された場合、これらの原料は腐敗状態に達した履歴があると判断できるかもしれない。
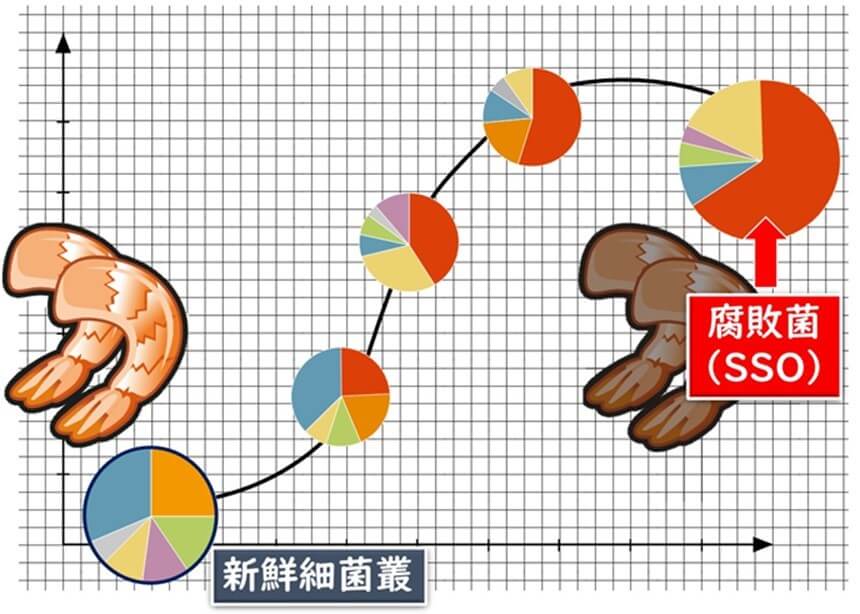
このような技術を活用することにより、その企業は原料を送り出す企業に対しての予防的警告の圧力をかけることも可能だ。
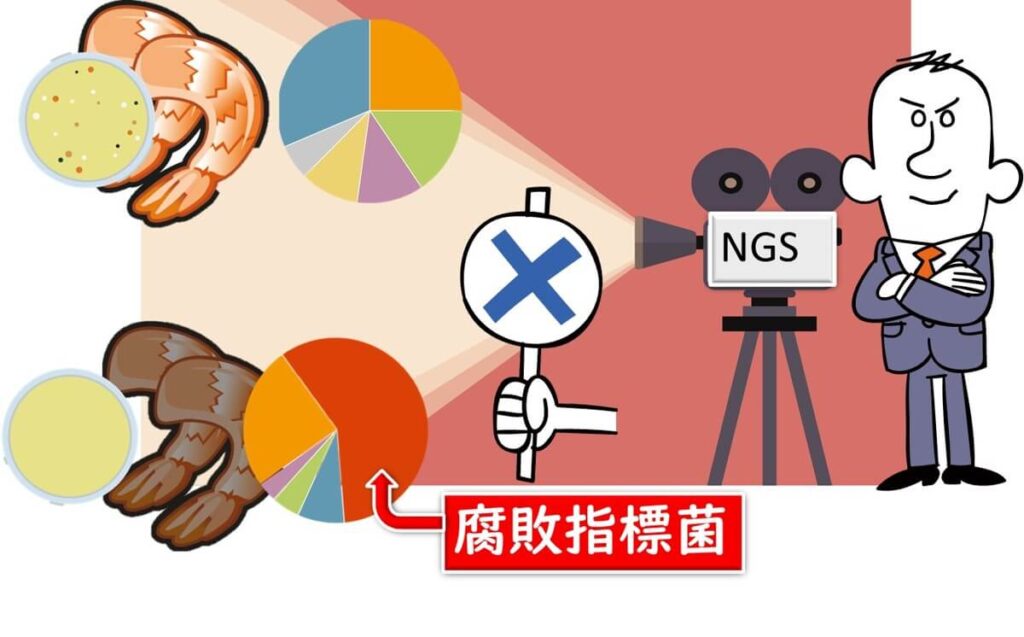
食品企業も、自社の製品の原料に対してこのような詳細な腐敗細菌叢の経時的変化のデータを蓄積することによって、ここで述べたような活用法も可能かもしれない。

食品微生物検査の使用における留意事項
以下に16SrRNAアンプリコンシーケンスを実施する上でのいくつかの留意点を述べておく。
生菌を検出できない
16SrRNAアンプリコンシーケンスの欠点の一つは死菌のDNAも検出してしまうことである。得られる食品中の菌叢から、微生物の生死は判定できない。食品中から DNAでなくRNAを抽出して逆転写酵素によってc-DNAを作成して16SrRNAアンプリコンシーケンスをする例もあるが、このような研究例は現時点では少ない。また、リボソーマル RNA は細菌の死後もかなりの長期間安定であることが知られているため、必ずしも RNA の逆転写酵素で解析する手法によって生菌を正確に検出できるとも限らない。したがって、現時点では、16SrRNAアンプリコンシーケンで得られる結果は死菌である可能性をを念頭に入れながら、得られる結果を解釈しなくてはならない。
※微生物が死滅した後もDNAが環境中に安定して存在する理由は、下記の別記事の【生菌を検出できない】の見出しで説明しているのでご覧ください。
PCR法による食品の微生物検査の留意点
全長解析でない
過去30年間、微生物の分離株の同定は、サンガー法による16 S ribosomal DNA 解析が盛んに行われてきた。 サンガー法でによる分離コロニーの同定は、基本的には全長解析が行われる。
一方、次世代シークエンサー( NGS)を用いて食品や環境から直接 DNA を抽出し解析をする16SrRNAアンプリコンシーケンスにおいては、16 S ribosomal DNA の部分配列 でOTUを推測する。
※OTUは何かについては別の記事でわかりやすくまとめているので、下記記事の16S rDNA 同定は OTU(operational taxonomic unit) に過ぎないをご覧ください。
遺伝子検査による微生物の同定
そもそも、NGSの主流技術はいずれも、その技術的な特性からショートリーディングの特性を持っている。つまり300から500ベースの短い断片しか解析できないという技術的限界がある。つまり、NGSによる16SrRNAアンプリコンシーケンスでは、16 S ribosomal DNA の全長を解析できないことだ。
※次世代シークエンサーのショートリーディングとは何か?なぜそうなるのかなどについての基礎事項については下記の記事をご覧ください。
DNAシーケンサーの原理をわかりやすく説明します
もちろん部分配列でのOTUであるので、種レベルで細菌叢のデータを得ることは難しい。 多くの場合は、属Genusレベルの同定か、もしくは、科Familyレベルの同定にとどまる。種レベルでも特定の種に最も近いという判定は可能であるが、あくまでも、その種に最も近い菌群ということに過ぎない。
また、ここで注意しなくてはならないのは、このような16 S ribosomal DNAの可変領域解析は、標的とする領域によって異なる結果が生じる可能性もあるということである。例えば、モックコミュニティ(あらかじめ既知の細菌の既知の割合で人工的に混合して作った細菌群集のこと)とモックコミュニティDNAを使用して、16SrRNAアンプリコンシーケンス結果に対するこれらDNA抽出方法、プライマー配列および、配列に用いる遺伝子領域の違いが、結果にどのように反映されるかを検討している研究例もある。異なるプライマー配列(V4 − V5、V1 − V2およびV1 − V2縮重プライマー)を用いると、それぞれに検出された属および種における相違が生じることが示されている。
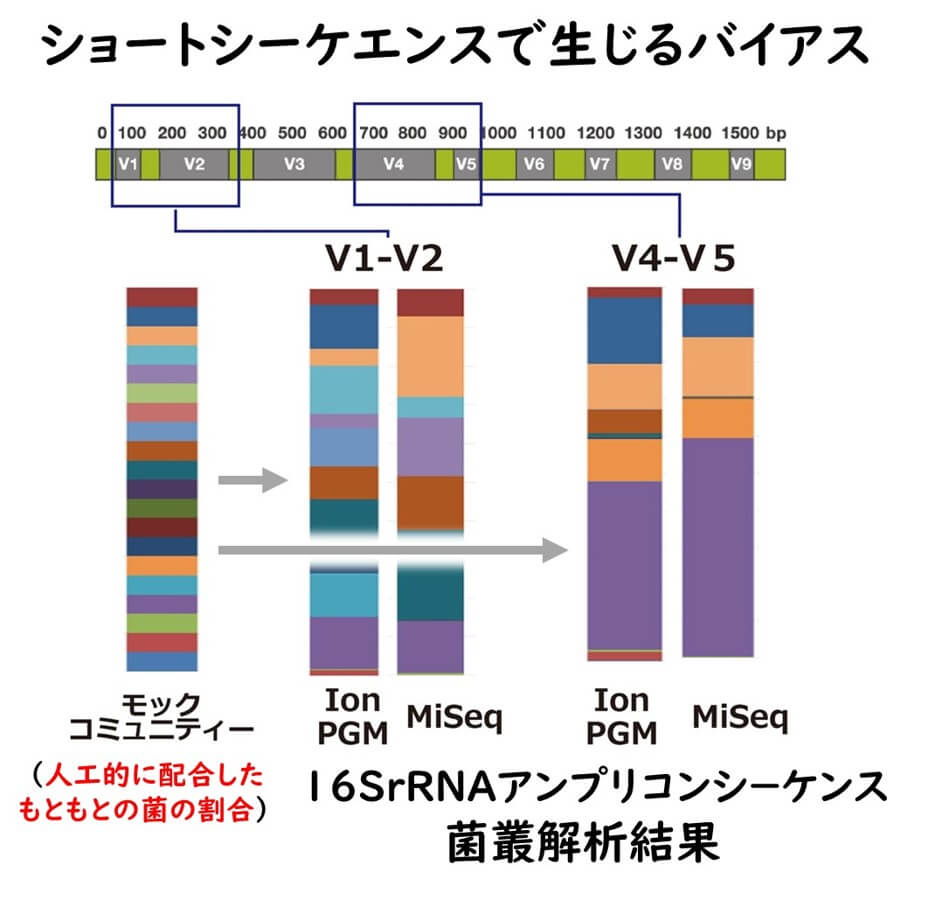
上の図は下記論文のデータを抽出し、このブログの著者が整理し直したものです。
Fouhy, F. et al.
16SrRNAgenesequencing of mock microbial populations-impact of DNA extraction method, primer choice and sequencing platform. BMC Microbiol. 16:123 (2016)
この論文はPubMed Central(PMC)で無料公開されています。
現在どの可変領域が最も正確に種レベルの同定に適してるかについての科学者の合意は得られてはいない。 少なくとも16 S ribosomal DNA の全長配列と比較すると、どの可変領域であっても、種の決定における解像度は低下し、また領域間によってその結果が異なるOTUとなる場合もあるということを留意しておく必要がある
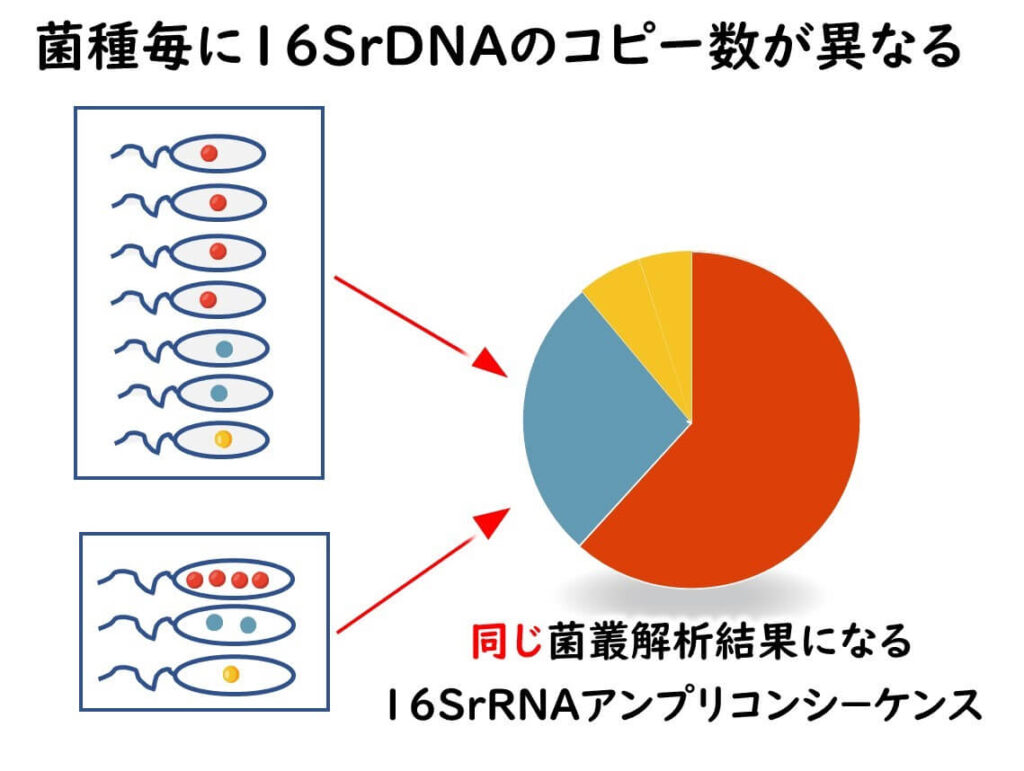
菌種によって16DNAコピー数が異なる
16Sr RNA遺伝子のコピー数が細菌によって異なる点も16SrRNAアンプリコンシーケンスにおけるバイアスの原因となる。細菌の16Sr RNA遺伝子のコピー数は1から15までのばらつきがある(コピー数は、Erythrobacter litoralisの1コピーからPhotobacterium profundumの15コピーまでさまざまである)参考文献)。 従って、16S-rRNA アンプリコン解析における環境試料中の16Sr RNA遺伝子の相対存在量は、細菌群集の細胞数の割合のみならず、それぞれの最近の持つ16Sr RNA遺伝子のコピー数によっても影響を受ける。
この事実にもかかわらず、これまでの多くの研究で、16Sr RNA遺伝子の存在量がそれらの配列を含む細菌の細胞としての存在比の代用尺度であると仮定している。しかし、このような仮定はやむを得えず便宜的に行っているものであり、正確性を欠くことを念頭に入れておく必要がある。
例えばある環境に4種類( 細菌 A,B,C,D)の微生物が存在していた場合を想定してみよう。それぞれの微生物が それぞれ1個ずつの16 S ribosomal DNA を保持している場合と、ある細菌Aが4コピーを、 細菌 Bが2コピーを、 細菌 Cが 1コピーを持っている場合では、16SrRNAアンプリコンシーケンスによって得られる細菌叢は異なるものとなる(図1)。16S-rRNA アンプリコン解析で得られる細菌叢のフローラの割合を解釈する場合には、このようなバイアスが生じているということを理解しておく必要がある。
絶対的定量ではない
16SrRNAアンプリコンシーケンスの微生物叢は絶対定量ではないということも留意しておく必要がある。
例えばここに2つのサンプルがあるとしよう。食品サンプルAには105 cfu/gの 細菌が存在していたとする。食品サンプルBには103 cfu/gの 細菌が存在していたとする。微生物の数の少ない食品サンプルBの16SrRNAアンプリコンシーケンスの結果を見てみよう。このサンプルではシュードモナスが優占種となっている。 一方食品サンプル A を見ると、シュードモナスは全体の割合からするとそれほどは多くない。ここで両方のサンプルの微生物の絶対数を無視して考えると、印象としては食品サンプルBにシュードモナスの割合が多いのでシュードモナスの代謝の影響が大きいと判断しまう危険性がある。しかし現実には食品サンプルAの方が絶対定量値はシュードモナスのほうが高い。従って実際には食品にシュードモナスが及ぼす影響は 食品サンプル A の方が大きい。
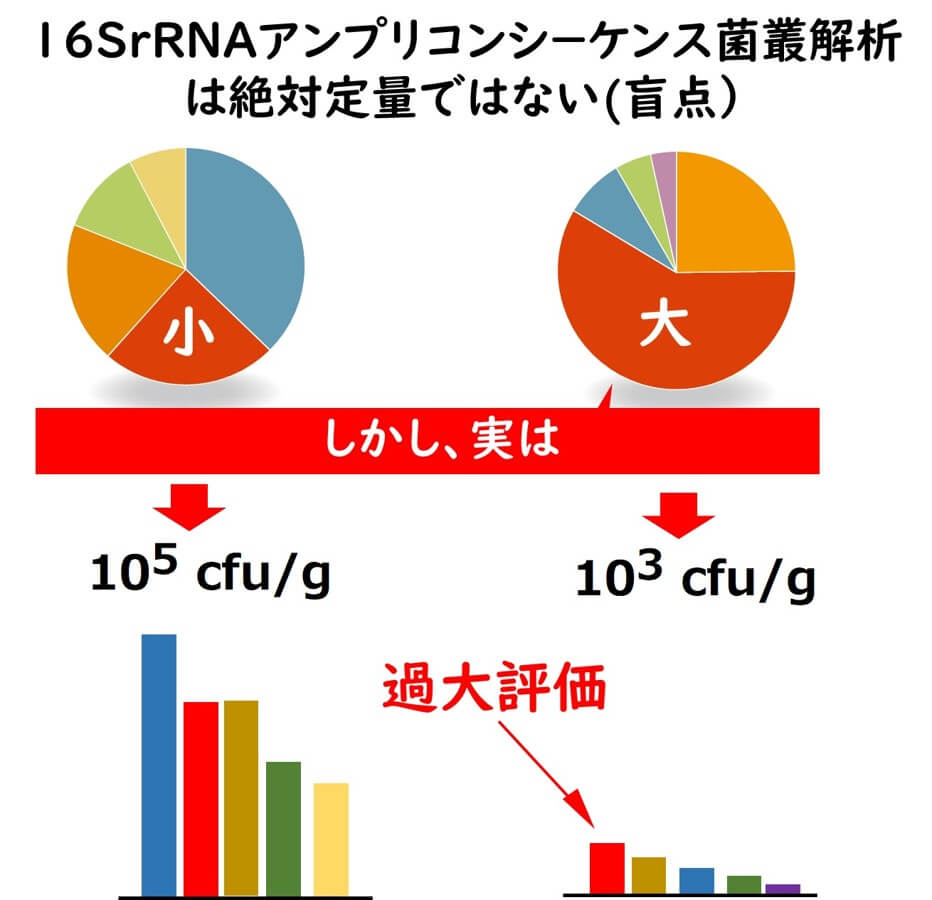
このように6S-rRNA アンプリコン解析で得られる微生物叢はサンプル中に存在している微生物の絶対数を無視して考えると、それぞれの微生物の果たす役割について誤った判断をしてしまう可能性がある。この点も留意しなくてはならない。
Sequence プロトコル,プライマーや遺伝子抽出法によるバイアス
16SrRNAアンプリコンシーケンスにおけるバイアスは標的とする領域のみに起因するわけではない。MiseqとIonPGMなどの異なる sequence のプロトコルによる結果の違いについてもいくつかの報告が出されている参考文献)。また、同じ領域を標的としていてもどのようなプライマーを用いるかによって結果が異なってくる参考文献)。16 S アンプリコンシーケンスを行う場合には、少なくとも用いているプライマーの特性を知っておくことと、むやみにプライマーを変更してサンプル間の微生物群集を比較することは避けるべきだろう。さらに、遺伝子の抽出キットの特性によっても異なる結果が出ることも多い参考文献)。
最近のレポートで、16SrRNAアンプリコンシーケンスにおいて異なるプロトコルで得られた結果を比較することは、まるで「りんごとみかんを比較するようなもの」であるとの指摘がされている参考文献)。りんごとみかんを比較するとは、全く異なるカテゴリーのものを比較することは意味がないという意味である(この表現は英語の慣用句でよく使われる)。

現時点で16S-rRNA アンプリコン解析における国際的に統一されたユニバーサル プロトコルというものは存在しない。したがって、16S-rRNA アンプリコン解析による微生物叢を実用的に活用していく際には、自社における配列決定に用いる配列領域やプライマー、遺伝子救出方法などのプロトコルを統一し、そのプロトコルを変更しないことが望ましいだろう。
とにかくまず使いはじめてみよう
自分の目的をしっかりと設定し、その目的を達成するためにこの方法を使いこなすという気持ちが大事であると考える。アカデミアの論文情報だけで、難しすぎるとあきらめないことだ。出版されている科学論文はアカデミアの人々が書くので完璧を求めたことを目指したテーマ設定や問題意識が多い。しかし食品産業界ではこれらの結果を待っているといつまでたってもこの技術を活用するに至らない。食品産業界では100%の正確性よりも迅速性が重要な場合が多い。このことを留意してこの技術を使っていくと良いのではないだろうか。
この活用法については食品産業界のそれぞれの現場において目的を設定して考えれば良い。とりあえず使って色々試行錯誤してみろというぐらいが良いのではないか?まずは、実験条件を定めた上で自社でプロトコルを統一し、その上で得られる結果は、属レベルであって種レベルであってもあくまでもOTUであることを割り切って、また生菌、死菌を割り切り、その上で自分たちの目的にどのように活用できるかを工夫してみるというのではないだろうか?このような活用を現場で行ってみて初めて色々な活用アイデアが出てくるものと考える。
